自己免疫疾患で重要なCD4+T細胞の変化を統一的に解析可能に
シングルセルメタアナリシスによる大規模自己免疫疾患プロファイリング
研究成果のポイント
概要
大阪大学大学院医学系研究科の安水 良明さん(博士後期課程)、森本 玲生さん(博士後期課程)、医学部医学科 竹内 大貴さん (学部5年)、大倉 永也特任教授(常勤) (基礎腫瘍免疫学)、免疫学フロンティア研究センター坂口 志文 特任教授(常勤)(実験免疫学)らの研究グループは、シングルセル解析を通じて詳細なCD4+T細胞のリファレンス構築と自己免疫疾患における大規模なプロファイリングを行いました。
シングルセルRNA-seqを用いて末梢血CD4+T細胞から18細胞種、12遺伝子プログラムを同定し、リファレンスを構築しました。このリファレンスにより、様々なデータセットにおいて詳細なCD4+T細胞プロファイリングを可能にします。このフレームワークを使用して、公共データを統合し、20疾患、953人、1,809,668 CD4+T細胞によるメタアナリシスを行い、多発性硬化症、重症筋無力症、全身性エリテマトーデスなどの自己免疫疾患に特異的なCD4+T細胞変化を同定しました。さらに、ゲノムワイド関連解析と統合解析を行うことで、CD4+T細胞遺伝子プログラムへの遺伝要因の集積の明らかにしました。
本研究ではCD4+T細胞の多様性と自己免疫疾患との関連を結びつけることに成功しました。このことは今後の自己免疫疾患治療の標的を考える戦略として役立つ可能性があります。さらに、メタアナリシスを通じてCD4+T細胞が疾患によって特徴的なプロファイルを示すことを明らかにしました。この結果はCD4+T細胞プロファイリングが自己免疫疾患の診断に応用される可能性を示しています。
本研究成果は、米国科学誌「Cell Genomics」に、1月4日(水)午前1時(日本時間)に公開されました。
図1. 本研究の概要
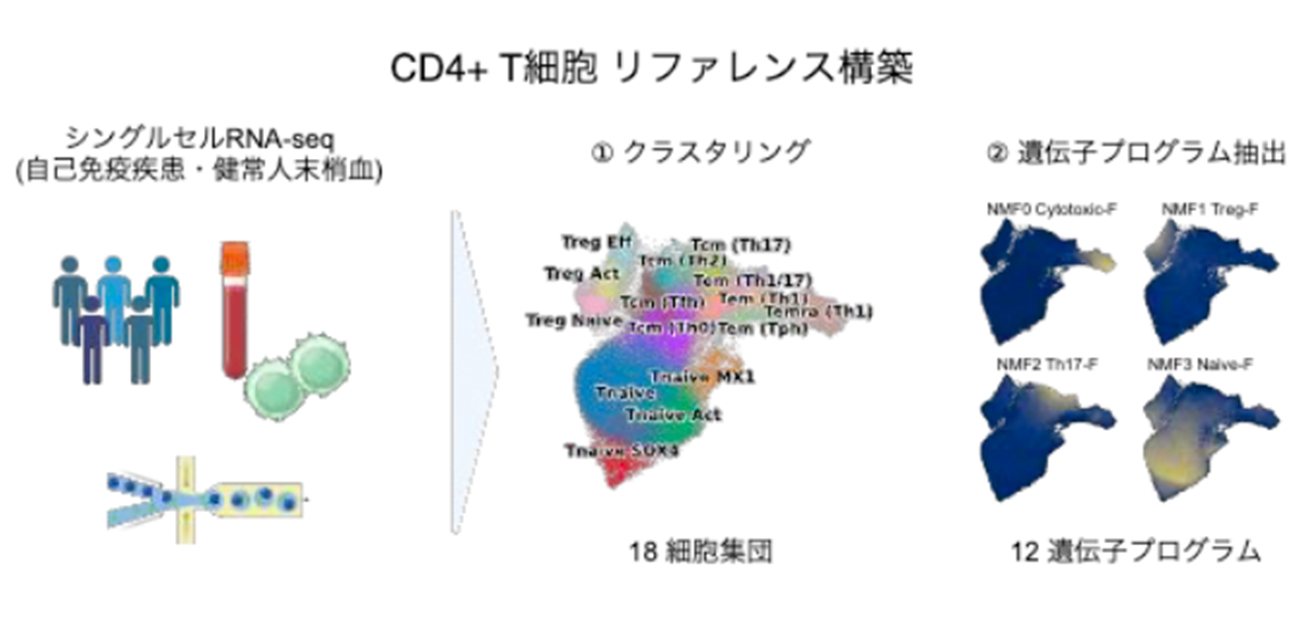
1. CD4+T細胞リファレンス構築
2. 自己免疫疾患プロファイリング を行った。
研究の背景
自己免疫疾患は免疫細胞が異常に自己に対して反応してしまうことで起きる疾患の総称で、多発性硬化症、重症筋無力症、全身性エリテマトーデス、関節リウマチを始め多くの疾患が含まれます。これまで、CD4+T細胞の異常が自己免疫疾患の原因となることが報告されていました。一方でCD4+T細胞はナイーブT細胞やメモリーT細胞などの状態やTh1, Th2, Th17, Tfh, Tregなどの細胞極性を有する多様な細胞集団であり、その複雑性のため、全体像が明らかになっていませんでした。また近年ではシングルセルRNA-seq解析によりその複雑性の一端は明らかになりつつありましたが、その解釈にはコンセンサスがありませんでした。そのため、CD4+T細胞のうち、どの要素が自己免疫疾患の発症に関わっているかは明らかになっていませんでした。そこで本研究は1) 多様なCD4+T細胞のリファレンスを構築し、2) その中でどの要素と各自己免疫疾患が関連しているかを明らかにすることを目的として行いました。
研究の内容
本研究ではまず、シングルセルRNA-seqを用いて末梢血CD4+T細胞のプロファイリングを行いました。まず、既存手法を用いて18個の細胞種を同定しました。そして、独自のパイプラインを構築することで、新たな末梢血シングルセルRNA-seqにおいて今回同定した細胞種分類を予測することを可能にしました。更に、CD4+T細胞はなだらかに変化する細胞集団であり、恣意的に境界を引きクラスタリングを行う従来の解析手法が適していなかったのではないかと考えました。そこで機械学習手法である非負値行列分解を適用することで、線引きをせずに連続的な遺伝子プログラムと細胞要素に分解しました。その結果、TregらしさやTfhらしさ、一型インターフェロン反応などからなる12の独立な遺伝子プログラムを同定しました。さらに、独自に開発したソフトウェア (NMFproj) により様々なバルクおよびシングルセルRNA-seqデータセットにおいて今回同定した12の遺伝子プログラムの強度を測定することを可能にしました。このリファレンスにより、様々なデータセットにおいて細胞分類による数的評価と遺伝子プログラムによる質的な評価を同時に行うことができるようになりました。
次に、このフレームワークを使用して、自己免疫疾患によるCD4+T細胞の数的・質的変化の評価を試みました。これまで多くの論文で自己免疫疾患における末梢血のシングルセルRNA-seq解析が行われてきましたが、一部の情報を使用するにとどまるなど、一貫性のない細胞分類による解析が行われていました。そこで、今回構築したフレームワークを使用して、公共データを含めた統合解析を行うことで疾患横断的な比較を通じた各自己免疫疾患のCD4+T細胞応答を抽出することができるのではないかと考えました。20疾患、953人、1,809,668 CD4+細胞によるメタアナリシスを行い、多発性硬化症、重症筋無力症、全身性エリテマトーデスなどの自己免疫疾患に特異的な数的・質的CD4+T細胞変化を明らかにしました。たとえば、多発性硬化症においてTh1やTh17細胞、重症筋無力症においてTh17細胞、全身性エリテマトーデスにおいてインターフェロン反応性ナイーブT細胞集団や活性化Tregなどが増加していることが明らかになりました。更に、機械学習手法を用いてCD4+T細胞プロファイルのみからある程度、自己免疫疾患が予測できることも明らかにしました。
最後に、ゲノムワイド関連解析と統合解析を行うことで、CD4+T細胞の各遺伝子プログラムへの遺伝要因の集積を明らかにしました。たとえば炎症性腸疾患や関節リウマチ、重症筋無力症などではTregらしさに関わる遺伝子プログラムへの遺伝的要因の集積が検出されました。他にも、Th17プログラムへの炎症性腸疾患、重症筋無力症、多発性硬化症との関連、一型インターフェロン反応と全身性エリテマトーデス、原発性胆汁性胆管炎、Th1プログラムと多発性硬化症との遺伝的要因の集積が明らかとなりました。本研究を通じて、自己免疫疾患において、遺伝的要因が様々なCD4+T細胞の特徴と関連していること、その表現型としてCD4+T細胞の数的・質的変化が生じていることが明らかとなりました。
本研究成果が社会に与える影響(本研究成果の意義)
本研究ではCD4+T細胞の多様性を明らかにし、今後の研究に使用可能なリソースを提供しています。このリソースにより、今後様々な疾患状態におけるCD4+T細胞変化を正確かつ詳細に捉えやすくなると考えられます。さらに、CD4+T細胞と自己免疫疾患との関連を細胞種単位・遺伝子プログラム単位で結びつけることに成功しました。このことは今後の自己免疫疾患治療の標的を考える戦略として役立つ可能性があります。さらに、メタアナリシスを通じてCD4+T細胞が疾患によって特徴的なプロファイルを示すことを明らかにしました。この結果はCD4+T細胞プロファイリングが自己免疫疾患診断に応用される可能性を示しています。
特記事項
本研究成果は、米国科学誌「Cell Genomics」に、1月4日(水)午前1時(日本時間)に公開されました。
タイトル: “Single-cell transcriptome landscape of circulating CD4+ T cell populations in autoimmune diseases”
著者名:Yoshiaki Yasumizu1,2,3,13, Daiki Takeuchi1,4,13, Reo Morimoto1,13, Yusuke Takeshima1, Tatsusada Okuno2, Makoto Kinoshita2, Takayoshi Morita5, Yasuhiro Kato5,6, Min Wang7,8, Daisuke Motooka3,9, Daisuke Okuzaki3,9, Yamami Nakamura1, Norihisa Mikami1, Masaya Arai1, Xuan Zhang8, Atsushi Kumanogoh3,5,6,10, Hideki Mochizuki2,3, Naganari Ohkura1,11, *, Shimon Sakaguchi1,12,*(*責任著者)
所属:
1. 大阪大学 免疫学フロンティア研究センター(IFReC) 実験免疫学
2. 大阪大学 大学院医学系研究科 神経内科学
3. 大阪大学 先導的学際研究機構(OTRI) 生命医科学融合フロンティア研究部門
4. 大阪大学 医学部医学科
5. 大阪大学 大学院医学系研究科 呼吸器・免疫内科学
6. 大阪大学 免疫学フロンティア研究センター(IFReC) 感染病態分野
7. Clinical Immunology Center, State Key Laboratory of Complex Severe and Rare Diseases, Peking Union Medical College Hospital, Chinese Academy of Medical Sciences and Peking Union Medical College, Beijing, China
8. Department of Rheumatology, Beijing Hospital, National Center of Gerontology, Institute of Geriatric Medicine, Chinese Academy of Medical Sciences, Beijing, China
9. 大阪大学 微生物病研究所 遺伝情報実験センターゲノム解析室
10.大阪大学 感染症総合教育研究拠点(CiDER)
11.大阪大学 大学院医学系研究科 基礎腫瘍免疫学
12.京都大学 再生医科学研究所 生体機能調節学
13.同等貢献
DOI:https://doi.org/10.1016/j.xgen.2023.100473
本研究は、日本学術振興会の特別推進研究「制御性 T 細胞による免疫応答制御の包括的研究」ならびに国立医療研究開発機構(AMED)の Leading Advanced Projects for medical innovation「制御性 T 細胞を標的とした免疫応答制御技術に関する研究開発」、大阪大学サイバーメディアセンターの大規模計算機システム公募方利用制度、大阪大学医学部 MD研究者育成プログラム、武田科学振興財団の協力を得て行われました。
参考URL
大阪大学免疫学フロンティア研究センター(IFReC) 実験免疫学
http://exp.immunol.ifrec.osaka-u.ac.jp/
用語説明
- CD4+T細胞
病原体感染時に抗原提示細胞から抗原提示を受けると増殖・活性化して、炎症を促進するサイトカインを産生して他の免疫細胞を活性化させる役目をもつ。
- メタアナリシス
複数の研究の結果を統合し、より高い見地から分析すること、またはそのための手法や統計解析のこと。
- RNA-seq
RNAをシークエンスすることで、遺伝子発現を網羅的に定量する手法。通常は多細胞を対象にするバルクRNA-seqを指すが、近年は1細胞ごとのRNA-seqを行うシングルセルRNA-seqも技術として確立してきた。
- ゲノムワイド関連解析
全ゲノム上で疾患・形質と関連する座位を網羅的に同定する手法。
