1細胞オミクスデータで X染色体不活化からの逃避を定量するソフトウェアを新開発
性差が生じるメカニズムの解明へ
研究成果のポイント
- 1細胞RNA-seqデータを用いて、X染色体不活化からの逃避を定量するソフトウェアsingle-cell Level inactivated X chromosome mapping (scLinaX)を新開発し、実装した。
- 複数の大規模1細胞RNA-seqデータセットにscLinaXを適用することにより、X染色体不活化からの逃避がリンパ球で特に強いことが判明
- X染色体不活化からの逃避がゲノムワイド関連解析における効果量の性差に寄与する可能性が示唆された。
概要
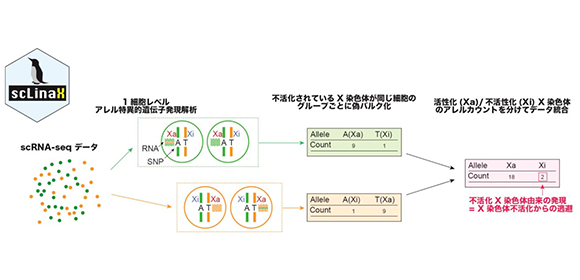
大阪大学大学院医学系研究科の友藤嘉彦 招へい教員(遺伝統計学/東京大学大学院医学系研究科 助教/理化学研究所生命医科学研究センター 客員研究員)、岡田随象 教授(遺伝統計学/東京大学大学院医学系研究科 教授/理化学研究所生命医科学研究センター チームリーダー)らの研究チームは、1細胞RNA-seqデータを用いて、X染色体不活化からの逃避を定量するソフトウェアsingle-cell Level inactivated X chromosome mapping (scLinaX)を開発・実装しました(図1)。X染色体不活化からの逃避は遺伝子発現の性差を生じさせる機序の一つであり、疾患や生命現象の性差の原因になっていると考えられています。研究グループはさらに、このソフトウェアを用いて、scLinaXを大規模なscRNA-seqデータセットや1細胞マルチオミクスデータセットに適用することで、X染色体不活化からの逃避がリンパ球において特に強いことを示しました。
scLinaXは、一般的な1細胞オミクスデータに幅広く適用可能なソフトウェアであり、統計解析ソフトウェアR上で使用可能なRパッケージとして公開されています(https://github.com/ytomofuji/scLinaX)。scLinaXは遺伝子発現や形質、疾患の性差の解明に資する基盤的手法となることが期待されます。
本研究成果は、2024 年7月31日(水)午前0時(日本時間)に米国科学誌「Cell Genomics」(オンライン)に掲載されました。
図1. scLinaXの概要
研究の背景
ヒトを含む哺乳類の場合、男性(オス)は1本のX染色体を、女性(メス)は2本のX染色体を持っています。この男女間のX染色体の本数の違いが遺伝子発現量に与える影響を補正するために、女性の2本のX染色体のうち、細胞ごとにランダムに決定された1本は不活性化され、遺伝子発現が殆ど起きない状態になります。残り1本だけが活性化された状態となるため、結果として男女間の遺伝子発現量が同じになるよう調節され、この現象を「X染色体不活化」と呼びます。しかし、X染色体不活化は時に不完全であり、X染色体上の遺伝子のうちおよそ12-20%については、不活化しているはずのX染色体からも部分的に遺伝子発現が起きることが知られています。この現象を「X染色体不活化からの逃避」と呼びます(図2)。X染色体不活化からの逃避が起きている遺伝子については、不活化しているX染色体からの遺伝子発現の分だけ、女性での遺伝子発現量が男性よりも高くなると考えられています。つまり、X染色体不活化からの逃避は遺伝子発現プロファイルの性差を生じさせます。しかしながら、生理的な条件下でX染色体不活化からの逃避を直接定量する技術がないために、X染色体不活化からの逃避の影響がどの細胞の遺伝子発現プロファイルにどれほどの影響を与えているのか、という点については解明されていませんでした。
図2. X染色体不活化からの逃避
研究の内容
研究チームは一般的な1細胞RNA-seqデータを用いて、X染色体不活化からの逃避を定量するソフトウェアsingle-cell Level inactivated X chromosome mapping (scLinaX)を開発・実装しました。scLinaXは、1細胞レベルのアレル特異的遺伝子発現解析データを元に、異なるX染色体が不活化している細胞同士を区別することで、X染色体不活化からの逃避を効率的に定量します。研究チームは、scLinaXを大規模な末梢血1細胞RNA-seqデータセットに適用することで、X染色体不活化からの逃避がミエロイド系よりもリンパ系の細胞において強いことを示しました(図3)。
図3. 各細胞種におけるDDX3X遺伝子のX染色体不活化からの逃避の強度。X染色体不活化からの逃避はリンパ球において強い
また、scLinaXをヒト多臓器1細胞RNA-seqデータセットに適用し、臓器単位・細胞種単位でX染色体不活化からの逃避の強度を定量しました。その結果、臓器単位の解析ではリンパ器官において、細胞種単位の解析ではリンパ球においてX染色体不活化からの逃避が強いことがわかりました(図4)。
図4. 多臓器scRNA-seqデータセットを用いたscLinaX解析。(A)臓器間比較(B)細胞種間比較。
さらに、1細胞マルチオミクス(RNA+ATAC)データセットに対するscLinaXの拡張として、scLinaX-multiを実装しました。scLinaX-multiを末梢血データセットに適用することで、クロマチン構造レベルでもX染色体不活化からの逃避がリンパ球において特に強いことがわかりました。
最後に、X染色体不活化からの逃避が複雑形質に与える影響を評価するために、男女間での層別化を行った上で、血球関連形質についてゲノムワイド関連解析(Genome-wide association study; GWAS)を実施し、GWAS効果量を男女間で比較しました。その結果、scLinaX解析でX染色体不活化からの逃避が検出された遺伝子である、PRKX遺伝子へのeQTL(expression Quantitative Trait Loci)効果を持つ遺伝子多型が、リンパ球数と関連を持っており、その効果量が男性よりも女性で大きいことがわかりました(図5)。この結果は、X染色体不活化からの逃避が遺伝子多型とヒト形質との関連の強さにも影響している可能性を示唆しています。
図5. リンパ球数GWASの効果量の性差。遺伝子多型rs6641874はT細胞・NK細胞においてPRKX遺伝子へのeQTL効果を有していた。
本研究成果が社会に与える影響(本研究成果の意義)
本研究で開発・実装されたscLinaXを用いることで、一般的な1細胞RNA-seqデータからX染色体不活化からの逃避を定量することが可能になりました。scLinaX及び本研究で構築されたX染色体不活化からの逃避の多臓器・多細胞種アトラスは遺伝子発現制御、形質、そして疾患の性差の解明に資する基盤となることが期待されます。
特記事項
本研究成果は、2024年7月31日(水)午前0時(日本時間)に米国科学誌「Cell Genomics」(オンライン)に掲載されました。
【タイトル】 “Quantification of escape from X chromosome inactivation with single-cell omics data reveals heterogeneity across cell types and tissues”
【著者】Yoshihiko Tomofuji1,2,3,4*, Ryuya Edahiro1,3,5, Kyuto Sonehara1,2,3,4, Yuya Shirai1,5, Kian Hong Kock6, Qingbo S. Wang1,3,4, Shinichi Namba1,4, Jonathan Moody7, Yoshinari Ando8, Akari Suzuki9, Tomohiro Yata1,10, Kotaro Ogawa10, Tatsuhiko Naito1,3, Ho Namkoong11, Quy Xiao Xuan Lin6, Eliora Violain Buyamin6, Le Min Tan6, Radhika Sonthalia6, Kyung Yeon Han12, Hiromu Tanaka13, Ho Lee13, Asian Immune Diversity Atlas Network, Japan COVID-19 Task Force, The BioBank Japan Project, Tatsusada Okuno10, Boxiang Liu14, Koichi Matsuda15, Koichi Fukunaga13, Hideki Mochizuki10, Woong-Yang Park12, Kazuhiko Yamamoto9, Chung-Chau Hon7, Jay W. Shin6,16, Shyam Prabhakar6,17,18, Atsushi Kumanogoh2,5,19, Yukinori Okada1,2,3,4,20,21*(*責任著者)
【所属】
1, 大阪大学大学院医学系研究科 遺伝統計学
2, 大阪大学先導的学際研究機構(OTRI) 生命医科学融合フロンティア研究部門
3, 理化学研究所 生命医科学研究センター システム遺伝学チーム
4, 東京大学大学院医学系研究科 遺伝情報学
5, 大阪大学大学院医学系研究科 呼吸器・免疫内科学
6, Genome Institute of Singapore (GIS), Agency for Science, Technology and Research (A*STAR)
7, 理化学研究所 生命医科学研究センター 遺伝子制御ゲノミクス研究チーム
8, 理化学研究所 生命医科学研究センター
9, 理化学研究所 生命医科学研究センター 自己免疫疾患研究チーム
10, 大阪大学大学院医学系研究科 神経内科学
11, 慶應義塾大学医学部 感染症学
12, Samsung Genome Institute, Samsung Medical Center
13, 慶應義塾大学医学部 呼吸器内科
14, Department of Pharmacy, National University of Singapore
15, 東京大学大学院新領域創成科学研究科 メディカル情報生命専攻
16, 理化学研究所 生命医科学研究センター 遺伝子制御回路研究チーム
17, Lee Kong Chian School of Medicine
18, Cancer Science Institute of Singapore
19, 大阪大学 免疫学フロンティア研究センター(IFReC) 感染病態
20, 大阪大学 免疫学フロンティア研究センター(IFReC) 免疫統計学
21, 大阪大学 ヒューマン・メタバース疾患研究拠点 (PRIMe)
本研究は、日本医療研究開発機構(AMED)ゲノム医療実現推進プラットフォーム事業・先端ゲノム研究開発(GRIFIN)「次世代ゲノミクス研究による乾癬の疾患病態解明・個別化医療・創薬」、免疫アレルギー疾患実用化研究事業「全ゲノム・一細胞シークエンス統合解析による関節リウマチの病態層別化と個別化医療実装」、ゲノム創薬基盤推進研究事業・ゲノム研究を創薬等出口に繋げる研究開発「ブレインアトラス創生による精神神経疾患のシングルセル・ゲノム創薬」(研究開発代表者:岡田随象)、の一環として行われ、大阪大学免疫学フロンティア研究センター、次世代主任研究者支援プログラム、大阪大学先導的学際研究機構、大阪大学大学院医学系研究科バイオインフォマティクスイニシアティブ、武田科学振興財団の協力を得て行われました。
用語説明
- 1細胞RNA-seq
個々の細胞から得られたRNA分子を鋳型として相補的DNA(cDNA)を作成し、その配列をハイスループットシークエンサーで決定する解析手法。個々の細胞ごとに遺伝子発現プロファイルを取得することができる。
- X染色体不活化
哺乳類のメスが持つ2本のX染色体のうち、1本が不活性化され、遺伝子発現が殆ど起きない状態になる現象。
- リンパ球
白血球の一種で、T細胞やB細胞などが含まれる。
- ゲノムワイド関連解析
(Genome-Wide Association Study; GWAS)ヒトゲノム全体にわたって存在する遺伝子多型と特定の疾患・形質との間の関連を網羅的に探索する手法。
- Rパッケージ
統計解析ソフトウェアRで使用される機能拡張モジュール。特定の解析を行うための関数群を含む。
- アレル特異的遺伝子発現解析
Allele-specific expression (ASE)解析。1塩基多型情報などを元に、RNA-seqで検出されたRNA分子が、対立遺伝子アレルのうち、どちらのアレルに由来するのかを決定する解析手法。
- ミエロイド系
血球細胞の一系統で、好中球、単球、マクロファージなどの細胞を含む。本研究では主に単球・樹状細胞が含まれていた。
- リンパ器官
リンパ球の生成、成熟、機能に関与する器官。リンパ節、脾臓、胸腺などが含まれる。
- 1細胞マルチオミクス(RNA+ATAC)
本研究においては、個々の細胞の遺伝子発現(RNA-seq)とクロマチンアクセシビリティ(ATAC-seq)とを同時に測定する解析手法を指す。
- クロマチン構造
本研究においては、ATAC-seqで評価可能なクロマチンアクセシビリティを指す。ATAC-seqでは、ヌクレオソームによって保護されていないDNA部位にトランスポゼースを使ってタグを挿入し、タグが挿入されたゲノム領域を増幅・配列決定することで、クロマチンアクセシビリティの情報を得る。
- eQTL(expression Quantitative Trait Loci)
遺伝子発現量に影響を与えるゲノム領域。
