より複雑で長いRNAの二次構造を高速に予測可能に
研究成果のポイント
- 必要な計算量を配列長の3乗から線形(1乗)に削減し、mRNAやウイルスRNAなどの長いRNA配列に対するシュードノットを考慮したRNA二次構造予測を可能にした。
- あらかじめ予測精度を見積もる指標を導入することで適切なパラメータが選択可能となり、二次構造予測の精度と汎用性が向上した。
概要
慶應義塾大学理工学部生命情報学科の佐藤健吾専任講師と大阪大学大学院医学系研究科の加藤有己助教からなる研究グループは、シュードノットと呼ばれる複雑なRNA部分構造を予測可能なRNA二次構造予測法(IPknot++)を開発することに成功しました。従来、数千塩基を超えるRNA配列に対するシュードノットを含めたRNA二次構造予測は計算量の観点から極めて困難でしたが、本手法はこれを克服して高速かつ高精度の予測を実現しました。メッセンジャーRNA(mRNA)やウイルスRNAなどの長鎖RNA配列においてシュードノットが関与する遺伝子発現制御機構の解明などへの応用が期待されます。
本研究成果は、2021年10月2日にイギリスの科学雑誌Briefings in Bioinformaticsのオンライン版に掲載されました。
研究の背景
RNA(リボ核酸)は生物の細胞内に存在し、DNAのゲノム(遺伝情報)からタンパク質の情報をコピーしている鎖状の高分子です。近年、COVID-19に対するワクチンで利用されるなど、その医療応用に向けた有用性が世界的に認知されています。このRNAの未知の機能を解明するには、強い相関関係がある構造の解析が不可欠で、コンピューターによりRNAの塩基配列データから、その折り畳み構造(RNA二次構造)を予測するアプローチは、時間とコストがかかる構造解析実験技術を補完する有力なツールとされています。
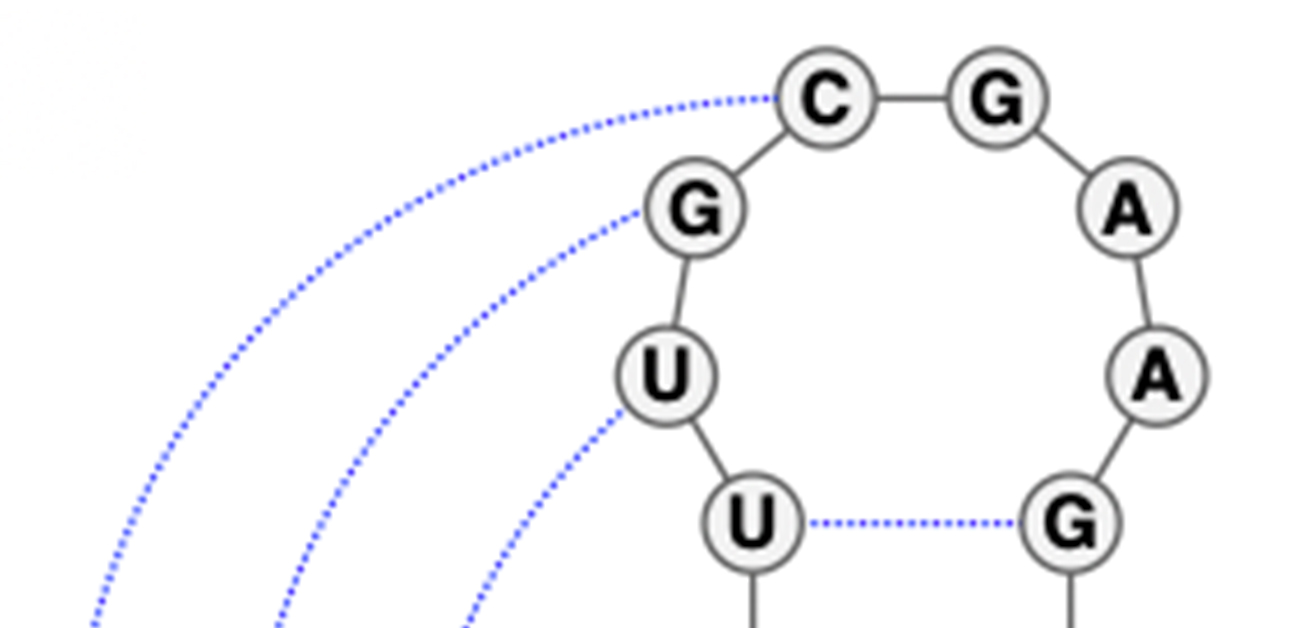
シュードノットは、RNA二次構造において重要なトポロジーの一つです(図 1)。任意のシュードノット構造を含むRNA二次構造予測の厳密解法は、その計算量(計算の複雑さ)が極めて複雑なクラスであることが証明されており、その実装は現実的ではないとされています。そのため、これまでの多くのRNA二次構造予測法はシュードノット構造を無視した予測を行います。しかしながら、シュードノット構造は翻訳やスプライシングの制御、リボソームのフレームシフトなどに関与することが知られており、シュードノット構造を考慮したRNA二次構造予測手法が求められています。
そこで、本研究グループは近似解法を用いて計算を高速化するIPknotを2011年に開発しました[Sato et al. 2011]。しかし、配列長に対して3乗に比例する計算時間を必要とする上に、配列長が500塩基を超えたあたりから予測精度が著しく低下するため、mRNAやウイルスRNAのような長い配列に適用することは困難でした。
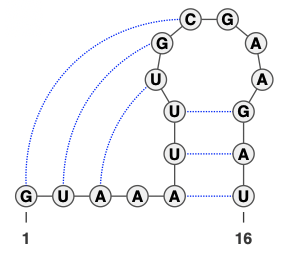
図1. シュードノット
研究内容・成果
本研究では、IPknotを改良することによって、mRNAやウイルスRNAのような長い配列に対しても高速かつ高精度でシュードノット構造を含むRNA二次構造を予測できる手法の開発を目指しました。高速化のために、ベースとなる計算モデルに解析手法高速化の点で良い近似を実現するLinearPartitionモデル[Zhang et al. 2020]を採用しました。LinearPartitionモデル自体はシュードノットを考慮した計算は行いませんが、これにIPknotによる近似解法を組み合わせることによって、配列長に対して線形(1次関数)の計算量でシュードノットを考慮したRNA二次構造予測を実現しました。さらに、あらかじめ予測精度を見積もる指標pseudo-expected accuracyをシュードノット構造に適用し、これに基づいて配列ごとに最適なパラメータを自動的に選択する方法を開発し、これによって高精度化を実現しました。
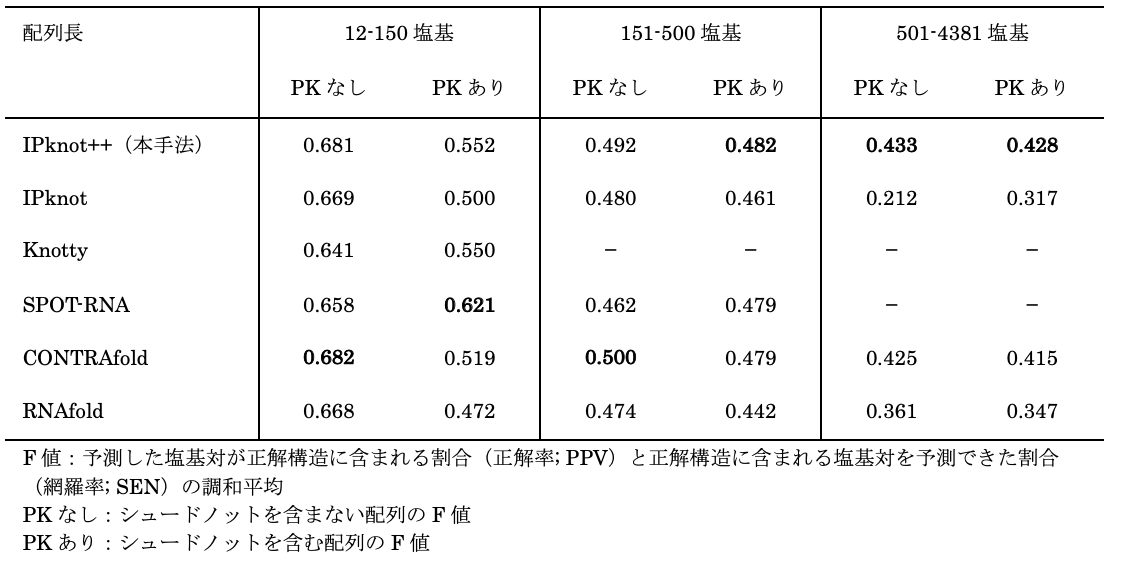
本研究で開発したIPknot++は、網羅的なベンチマークにおいて幅広い条件でシュードノットを含まないRNA二次構造予測と同等の計算速度でありながら(図 2)、シュードノットを含む配列に対しても良好な予測精度であることを示しました(表 1)。
本手法IPknot++はウェブサイトよりダウンロード可能なほか、ウェブサーバとして簡単に利用することができます。
ダウンロード用ウェブサイト https://github.com/satoken/ipknot/
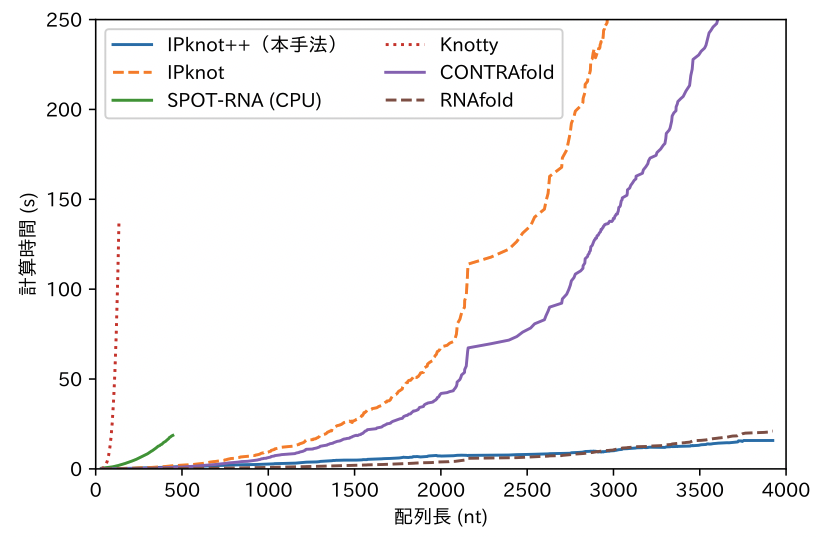
図2. 他手法との計算時間の比較
表1. 配列長ごとの他手法とのF値の比較
今後の展開
RNA二次構造予測は計算機によるRNA配列解析において最も基盤となる技術です。本研究で開発したIPknot++は、長い配列に含まれるシュードノット構造を予測できることを示しました。シュードノット構造は翻訳やスプライシングの制御などに関与することが知られています。特に近年、SARS-CoV-2でリボソームのフレームシフトを引き起こす要因となっていることがわかり、創薬ターゲットとしても注目されています。このようなRNAの機能を推定するために、本手法により実現した長い配列に対するシュードノットを考慮したRNA二次構造予測が有用であると期待されます。
特記事項
<原論文情報>
Sato, K., Kato. Y. “Prediction of RNA secondary structure including pseudoknots for long sequences.” Briefings in Bioinformatics.
doi: 10.1093/bib/bbab395
<参考文献>
[Sato et al. 2011] Sato, K., Kato, Y., Hamada, M., Akutsu, T. & Asai, K. “IPknot: fast and accurate prediction of RNA secondary structures with pseudoknots using integer programming.” Bioinformatics 27, i85–i93 (2011)
[Zhang et al. 2020] Zhang, H., Zhang, L., Mathews, D. H. & Huang, L. “LinearPartition: linear-time approximation of RNA folding partition function and base-pairing probabilities.” Bioinformatics 36, i258–i267 (2020)
用語説明
- RNA二次構造
立体構造の骨格を成す塩基対の集合で、A-U、G-C、G-Uの規則で結合する。