シングルセル高精度比較解析アルゴリズムを開発
細胞時系列比較により疾患関連遺伝子を同定する新技術
研究成果のポイント
- 細胞集団が時間的に複雑に変化する過程をシングルセルレベルで比較できる新規アルゴリズムを開発
- これまで比較すべき細胞集団において、枝分かれのない時系列を個別に選択する必要があったが、効率の良い分岐構造の比較法を実装することで、高精度の比較解析が一度に可能に
- コンピュータでグローバルな細胞時系列データを比較することにより、事前知識を用いた手法では見つからないような疾患関連遺伝子が同定され、医療への応用に期待
概要
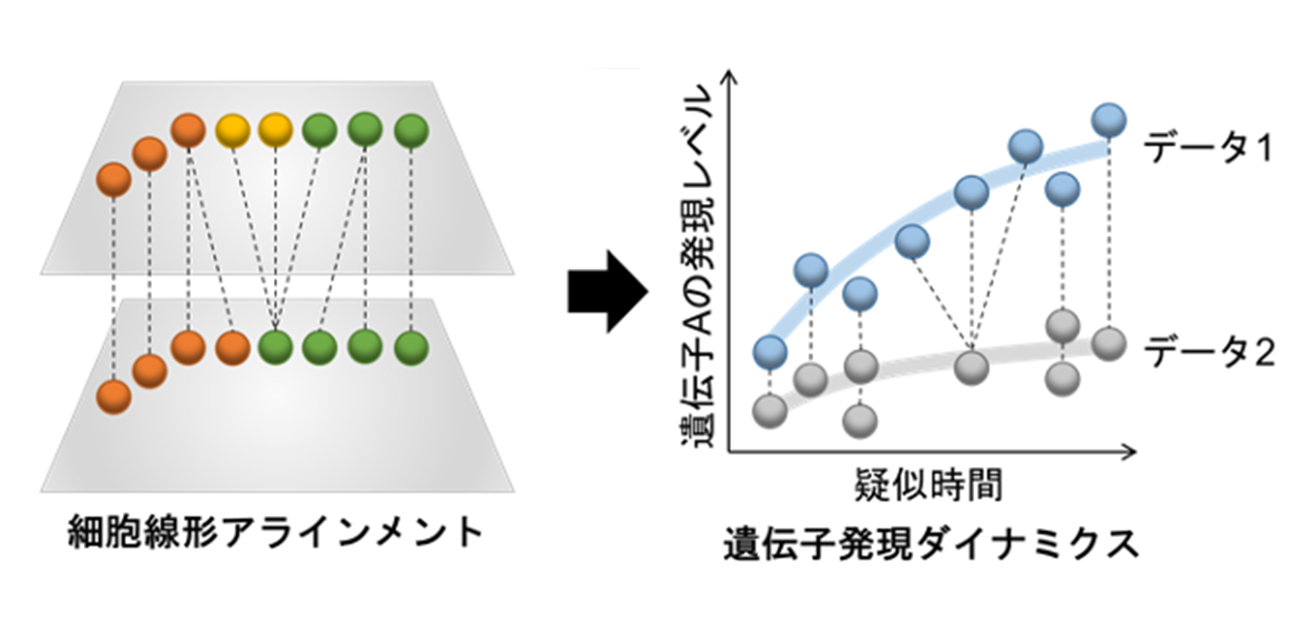
大阪大学大学院医学系研究科の加藤有己准教授、大学院生の杉原礼一(博士課程)(神経遺伝子学)らの研究グループは、様々な経過時間の細胞が含まれる遺伝子発現データセットのペアに対し、疑似的な時系列 (疑似時間) に沿って分岐を含めてシングルセル比較解析を行う計算手法CAPITALを開発しました(図1)。
これまで、細胞の分化など多くの場合、分岐を含む細胞動態を解析するための疑似時系列比較は、解析者が事前知識を使って直線の経路を1つずつ選択する必要があり、コンピュータによる自動化は実装さ れていませんでした。
今回、研究グループは、計算機科学の分野で提案されていた木のアラインメントを適用することにより、分岐のある木構造に対する疑似時系列シングルセル比較解析を行うツールCAPITALを開発しました。これにより、比較すべき最適な分化経路がアルゴリズムにより決定されるため、精度向上が見込まれます。例えば、データ1としてある疾患患者の、そして、データ2として健常者の遺伝子発現データを取り、CAPITALを実行することで、これまで検出できなかった疾患関連遺伝子の同定につながり、医療分野への応用が期待されます。
本研究成果は、英国科学誌「Nature Communications」に、10月14日(金)18時(日本時間)公開されました。
図1. 分岐を含む細胞集団の疑似時系列比較アルゴリズム
研究の背景
生体組織や器官はさまざまな細胞の集団から構成されていますが、それらで発現している遺伝子の種類や量を細胞1つ1つに分解して調べることができるようになりました。細胞は分化という過程を経て最終的な形になり、それぞれの機能を発揮しますが、遺伝子発現の経時変化を確認することが重要です。例えば、正常なマウスと病気のマウスから取得したシングルセル遺伝子発現データセットのペアを比較することで、病気を制御する遺伝子は何かといった疑問を、コンピュータを使って解明することができると考えられます。
細胞分化には時間軸に沿って枝分かれする過程が含まれることが多く、これまでのシングルセル比較解析では、解析者が事前知識を使って1つの直線の経路を選択する必要がありました。もちろん、すべての組み合わせをしらみつぶし的に調べ、最適な組み合わせを選べばよいのですが、計算時間がかかってしまいます。一方、互いに関連のある2つのシングルセルデータセットを先に統合し、互いのデータに共通の分化経路を計算することで、分岐を選択する問題を回避することも可能ですが、統合により元の経路情報が適切に保存されない可能性がありました。
研究の内容
加藤准教授らの研究グループでは、遺伝子発現が類似している細胞部分集団(クラスター)を要素とした木構造の経路をあらかじめ計算し、それらをアラインメントするアルゴリズムにより、シングルセル比較解析ツールCAPITAL (comparative analysis of pseudotime trajectory inference with tree alignment)を開発しました。分岐が複数箇所出現するように設計された2,278個の合成データセットのペアを用いて、先述のデータ統合手法による複数の最新手法と網羅的に比較したところ、CAPITALが統計的に有意に経路 (時系列) 情報を保存してアラインメントできることを示しました(図2)。
次に、異なる研究で得られたヒトの骨髄細胞の遺伝子発現データのペアに対し、CAPITALを用いてシングルセル比較解析を実施しました。当該データには造血幹細胞から赤血球や単球など分化した細胞までが含まれ、時間軸に沿って分岐を多く含むことが知られています。CAPITALによるシングルセル比較解析により、たとえデータが異なっていても、互いに同じ細胞型へのアラインメントに成功しました。また、代表的なマーカー遺伝子の発現ダイナミクスが類似していることを確認しました。
さらに、ヒトとマウスといった異なる生物種から得られた骨髄細胞データに対してCAPITALによるシングルセル比較解析を行い、進化の観点から見た細胞動態の違いを示唆する結果が得られました(図3)。
図2. CAPITALは最新のデータ統合手法よりも時系列情報を保存する
図3. 造血幹細胞から赤血球への分化経路におけるヒトとマウスの遺伝子CSF1の発現の違い
本研究成果が社会に与える影響(本研究成果の意義)
本研究成果により、細胞動態をシングルセルレベルで比較する上で、分岐を含む複雑な分化経路を扱うことが可能となりました。例えば、血液など複雑に分化することが知られている細胞集団に焦点を当て、データ1としてある疾患患者の、データ2として健常者の遺伝子発現データを取り、CAPITALを適用することで、これまで検出できなかった疾患関連遺伝子の同定につながり、医療分野への応用が期待されます。
特記事項
本研究成果は、2022年10月14日(金)18時(日本時間)に英国科学誌「Nature Communications」(オンライン)に掲載されました。
タイトル:“Alignment of single-cell trajectory trees with CAPITAL”
著者名:Reiichi Sugihara1, Yuki Kato1,2,*, Tomoya Mori3 and Yukio Kawahara1,2 (*責任著者)
所属:
1. 大阪大学 大学院医学系研究科 ゲノム生物学講座 神経遺伝子学教室
2. 大阪大学 先導的学際研究機構 生命医科学融合フロンティア研究部門
3. 京都大学 化学研究所 バイオインフォマティクスセンター
DOI:https://doi.org/10.1038/s41467-022-33681-3
参考URL
加藤有己 准教授 研究者総覧
https://rd.iai.osaka-u.ac.jp/ja/f5271613ba0be57f.html
用語説明
- 疑似時間
生体組織や器官から細胞を採取すると、それ以降完全に同じ細胞を採取できないため、実時間の追跡は不可能である。そこで、同じ組織から採取した細胞集団には様々な時間経過の細胞が含まれると仮定し、それらに対応する疑似的な時間を疑似時間という。
- シングルセル比較解析
ある細胞集団を構成する個々の細胞の遺伝子発現量などを観測したデータを使い、互いに関連のあるシングルセルデータ同士を比較すること。例えば、2つのデータの間で発現量に差のある遺伝子を同定するといった解析を含む。
- 分化
遺伝子発現の変化により、細胞が特定の細胞型に変化していく過程。
- 木のアラインメント
比較する2つの木構造において、親子関係などの階層構造を考慮しながら、それぞれを構成する素(本研究における細胞クラスター) 同士の対応付けを取ること。