AlphaFold(アルファフォールド)を用いて タンパク質間の新しい結合の発見を実現
AI技術が駆動する新たな実験科学の可能性
研究成果のポイント
- ノーベル賞を受賞したAIプログラム「AlphaFold(アルファフォールド)」を用いて、大規模なタンパク質間の結合スクリーニングを行い、新しい結合を発見
- AlphaFold2プログラムをスーパーコンピュータ上で運用し、迅速に結合スクリーニングを行うことによって、実験の負担を低減することが可能に
- AI技術が実験科学にもたらす新しい可能性が、インフォマティクスと実験科学のギャップを埋めて、生命科学の新たな発見を推進することに期待
概要
大阪大学大学院生命機能研究科の河口真一助教、大学院生の徐 鑫さん(博士課程)、甲斐歳恵教授らの研究グループは、米国DeepMind社によって開発・公開されているAIプログラムAlphaFoldを用いて、タンパク質間の結合をスーパーコンピュータ上で大規模に予測し、結合タンパク質のペアを新しく見出しました。
タンパク質間の結合は、タンパク質の機能を発揮させるため、あるいは機能を制御するために、最も重要な性質の1つです。そのため、着目するタンパク質と結合するタンパク質を探索する試みが、頻繁に行われています。しかしながら、結合タンパク質の探索には、時間と労力がかかることが課題でした。
今回、研究グループは、AlphaFold2プログラムを用いて、コンピュータ上で迅速にタンパク質間の結合スクリーニングを行うことによって、結合する可能性のあるタンパク質パートナーを絞り込み、これまでに直接の結合が知られていなかったタンパク質ペアを見出しました。
この結果は、AI技術が実験科学に対して新たな可能性を提供できることを示しており、AIを用いるインフォマティクスと実験科学のギャップを埋めて、生命科学の新たな発見を推進すると期待されます(図1)。
本研究成果は、国際科学誌「eLife」に、4月22日(火)15:30(日本時間)に公開されました。
図1. 革新的なAI技術が、インフォマティクスと実験科学の架け橋となり、相乗的な発展が期待される。
研究の背景
生体内では、約1〜2万種類ものタンパク質が相互作用することによって、複雑な生体反応を担っています。そのため、タンパク質の機能を研究する場合には、それと相互作用する他のタンパク質を探索することが行われます。しかしながら、実験的に相互作用を解析するには、多大な時間と労力が必要であり、より迅速で簡便な方法が求められていました。そのような状況の中で、タンパク質の立体構造を予測するAlphaFoldプログラムが開発・公開されました。さらに、AlphaFoldがタンパク質間複合体の構造も予測できる可能性があることから、結合タンパク質の探索に有効ではないかと期待されていました。
研究の内容
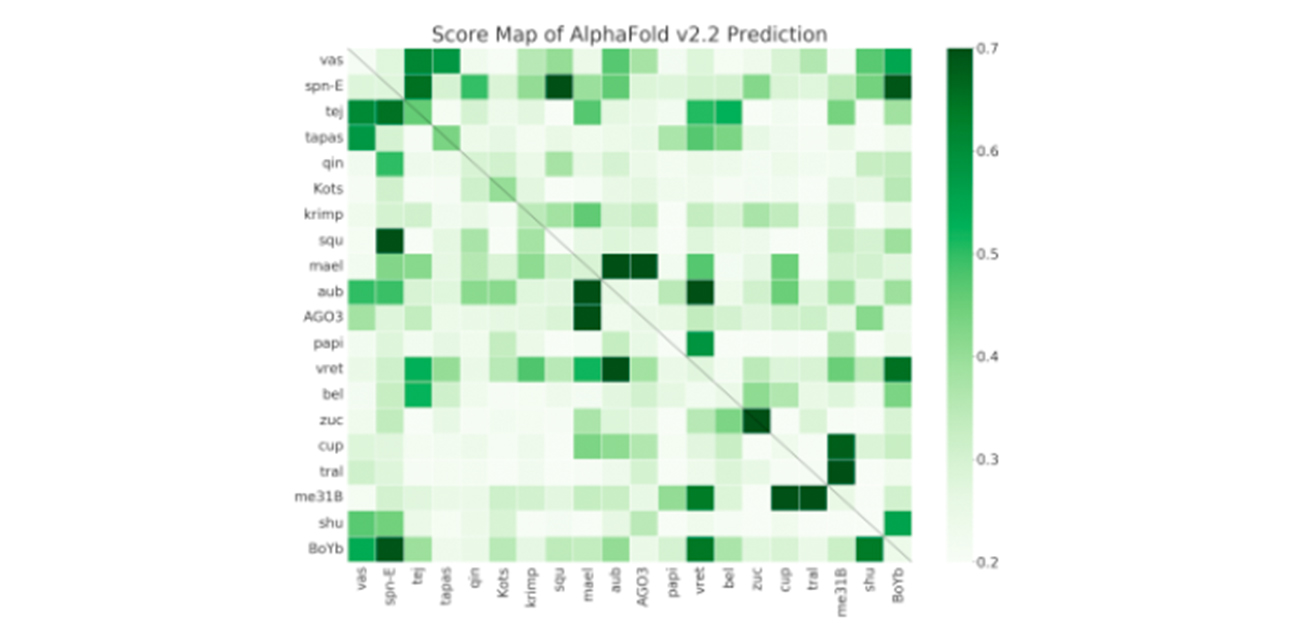
甲斐教授らの研究グループでは、ヌアージュという細胞内顆粒に着目し、その構造と機能に関する研究を行っています。本研究では、ヌアージュ顆粒に局在することが知られている20種類のタンパク質について、それらが直接に結合する可能性を、AlphaFold2プログラムを用いて予測しました。2つのタンパク質が1:1で結合することを仮定して、20×20=400ペアの予測計算を行いました(図2)。その結果、これまでに知られているタンパク質に加えて、新規なタンパク質複合体の候補が示されました。実際に、3つのタンパク質ペアについて、培養細胞内で発現させた2つの候補タンパク質が結合することがわかりました。さらに、SquとSpnEという2つのタンパク質ペアについては、予測された複合体の立体構造(図3)を基に、複合体形成に重要なアミノ酸残基を推定しました。実際に、これらの残基を変異させたSqu変異体は、SpnEタンパク質と結合しませんでした。このことから、予測された複合体の立体構造は確からしいと考えられます。
このスクリーニングをさらに拡張し、Piwiと呼ばれるタンパク質と、細胞内に存在する約1万種類のタンパク質との結合スクリーニングを行いました。その結果、これまでに報告されていない新規な結合タンパク質の候補を見出すことができました。
本研究では、AlphaFold2の大規模計算を行うため、大阪大学のスーパーコンピュータ(SQUID)を用い、並列計算化することによって、迅速に予測スクリーニングを行うことができました。
図2. 20種類のヌアージュタンパク質の複合体スクリーニングを総当たりで行った。ヒートマップの濃い緑は、予測された信頼性スコアが高いことを示す。
図3. AlphaFold2で予測されたSpnEタンパク質とSquタンパク質の複合体の立体構造モデル。SpnEの表面にSquの一部が埋まっており、3つの残基が結合に重要であることが示された。
本研究成果が社会に与える影響(本研究成果の意義)
本研究成果により、タンパク質間の結合を迅速に探索することが可能になり、タンパク質の機能解析を加速できることが期待されます。また、コンピュータ上のスクリーニングでは、実験的制約を受けないことから、特に毒性の強いタンパク質に対する予測が、研究を進める上で有効になると期待されます。がん細胞では、特にがんの原因と推定されるタンパク質が過剰に発現される場合があり、正常細胞と比べて、より多くの結合が生じていると考えられます。その中には、細胞にとって好ましくないタンパク質間結合も含まれているのではないかと推定されます。この例のように、AI科学を含むバイオインフォマティクスが、実験科学を新たな方向へと進展させることが期待されます(図1)。
特記事項
本研究成果は、2025年4月22日(火)15:30(日本時間)に国際科学誌「eLife」(オンライン)に掲載されました。
タイトル:“In silico screening by AlphaFold2 program revealed the potential binding partners of nuage-localizing proteins and piRNA-related proteins”
著者名:Kawaguchi Shinichi, Xu Xin, Soga Takashi, Yamaguchi Kenta, Kawasaki Ryuuya, Shimouchi Ryota, Date Susumu, and Kai Toshie
DOI: https://doi.org/10.7554/eLife.101967.
なお、本研究は、武田科学振興財団 (J191503009), 日本学術振興会科学研究費助成事業 (21H05275、20H05390)および大阪大学データビリティフロンティア機構 IDS学際共創プロジェクト(Na22990007)の支援を受けて行われました。また、大阪大学 D3センター 伊達進教授、曽我隆特任准教授(常勤)、及びNECソリューションイノベータ株式会社の協力を得て、大阪大学D3センターのスーパーコンピュータ(SQUID)で、AlphaFold2の高速計算が可能になりました。
参考URL
用語説明
- AlphaFold(アルファフォールド)
米国DeepMind社が開発したAIプログラムで、タンパク質のアミノ酸配列から、その立体構造を予測する。2024年にノーベル化学賞を受賞している。タンパク質単体の立体構造だけでなく、複数のタンパク質からなる複合体の立体構造も予測する。
- タンパク質の立体構造
タンパク質は20種類のアミノ酸配列が、遺伝情報によって設計された順番に繋がっているポリマーである。アミノ酸には、親水性なものと疎水性なものがあり、それらの並び方に特有の三次元構造をとる。立体構造表面の化学的特性は、他の分子との結合に重要な因子である。
- ヌアージュ
ショウジョウバエの生殖細胞の核膜近傍に存在する顆粒で、多くの種類のタンパク質やRNAが局在している。piRNAと呼ばれる小分子RNAを産生する場所であり、ゲノムDNAの安定性に重要である。
- Piwi
piRNAと結合して、ゲノムDNAに損傷を引き起こす可能性のある転移因子の発現を抑制する。
- バイオインフォマティクス
ゲノムDNAやタンパク質のアミノ酸配列に含まれる特徴を解析し、機能を推定する。また、生体分子の検出技術が向上しており、多くの分子を網羅的に、定量的に解析できるようになってきた。そこから得られる大量のデータを解析し、生物学的な特徴を解析する。
