タンパク質の見えなかった部分を見る
遺伝子発現スイッチのオンオフ機構を活写する
研究成果のポイント
実験的に観測が難しく、その振る舞いが不明であった天然変性領域の振る舞いをコンピューターシミュレーションと生化学機能実験によって明らかにしました。
概要
決まった構造をとらず、常に形を変えていると考えられている天然変性領域と呼ばれるタンパク質領域に、いま世界的に注目が集まっています。その理由は、タンパク質機能を調節している領域の多くがこの「天然変性領域」であるからです。これまで、タンパク質は決まった構造をとって機能を発揮していると考えられてきましたが、「天然変性領域」はこのルールに明らかに反しているように見えます。では決まった構造をとらないにもかかわらず、なぜタンパク質の機能を制御できるのでしょうか。立命館大学の笠原浩太助教、大阪大学の肥後順一特任教授(常勤)、中村春木教授、および横浜市立大学の椎名政昭助教、緒方一博教授らの研究グループは、タンパク質の天然変性領域の翻訳後修飾 (リン酸化)によってタンパク質機能のON/OFFのスイッチが切り替わる機構について、コンピューターによる構造シミュレーションと生化学実験による機能・構造解析によって、原子レベルで明らかにしました。今回の私たちの研究の結果、天然変性領域は、実は自由に形を変えているのではなく、リン酸化の有無に応じて、限定された種類の構造(特異的構造)を変換することで、タンパク質の分子スイッチとして機能していることが明らかになりました。
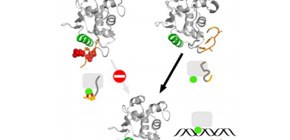
図1
細胞の分化を調節するEts1という転写因子の構造を示しています。オレンジ色で示している部分が「天然変性領域」で、リン酸化を受けている状態(上左)ではリン酸部分を赤色で示しています。また緑色で示した部分が DNA に結合する領域で、下の図はDNAに結合した状態を示しています。Ets1はリン酸化を受けることで天然変性領域の形が変わり、DNAに結合する領域を覆うようになることがわかります。このようにリン酸化によって天然変性領域が形を変えることで、Ets1がDNAに結合できる状態からDNAに結合できない状態にスイッチされることが明らかになりました。
研究の背景
人体は様々な組織によって構成され、各組織は機能分化した細胞によって形成されています。例えば、神経細胞は、神経の機能に必要なタンパク質を、筋細胞は、筋肉の機能に必要なタンパク質を合成します。各細胞には、その組織の機能に必要なタンパク質の情報を遺伝子から選択的に読み取り、合成する仕組みが備わっています。この情報を読み出すスイッチのON/OFFを司るのが、転写因子というタンパク質で、DNAに結合して遺伝子の読み出しを司令します。今回、我々が注目したのは、血液細胞の機能にとって重要なタンパク質合成を制御するEts1という転写因子です。Ets1の異常は、がんの浸潤・転移に関与すると報告されており、臨床的にも重要なタンパク質です。
タンパク質の機能が発揮されるためには、特異的な立体構造の形成が重要であると考えられています。しかし、ヒトを始めとした高等生物のタンパク質には、特定の構造を持たない天然変性領域が多く含まれます。天然変性領域は、翻訳後修飾(化学修飾)を受け、それが生体内ではあたかもスイッチのように機能し、タンパク質機能が調節されることが分かっていました。Ets1にも天然変性領域が含まれており、リン酸化されるとEts1はDNAに結合しなくなります(スイッチOFF)。しかし、スイッチとして機能する天然変性領域は、従来の実験方法では特定の構造が検出できず、その振る舞いは謎に包まれていました。
研究の内容
天然変性領域は従来の実験による構造決定法では解析が難しいため、立命館大学の笠原浩太助教、大阪大学の肥後順一特任教授(常勤)、中村春木教授らが独自のシミュレーションプログラムmyPrestoを用い、Ets1の天然変性領域の振る舞いをシミュレーションで明らかにしました。続いてシミュレーション結果を基に、横浜市大では、天然変性領域に半網羅的にアミノ酸置換を導入して試験管内でリン酸化し、Ets1のDNA結合を速度論的に解析しました。その結果、天然変性領域がリン酸化されると、DNAとの結合部分をふさぐ構造が現れ、DNA 結合が阻害されることがわかりました。従来、特定の構造を形成しないと考えられていた天然変性領域が化学修飾を受けることによって限定された種類の構造を形成することが、天然変性領域の翻訳後修飾によるスイッチ機構であることが明らかになりました。
今後の展開
本研究は、Ets1の天然変性領域が、自身の翻訳後修飾の状態に応じて、異なる構造を形成することで機能することを明らかにしました。これまで、実験によって構造を決定できない領域は、創薬の標的とされてきませんでしたが、今回の研究成果によって、実験的に構造を決定できない天然変性領域についても創薬の標的となりうる可能性が拓かれました。今後は、疾患の発症・進展に関与する他のタンパク質の天然変性領域についても解析し、創薬応用の観点からも分子機構の研究を進めていく予定です。
特記事項
本研究は、英科学誌『Nucleic Acids Research』に掲載されました。(英国1月4日付オンライン)
掲載論文:
Phosphorylation of an intrinsically disordered region of Ets1 shifts a multi-modal interaction ensemble to an auto-inhibitory state
Kota Kasahara * , Masaaki Shiina * , Junichi Higo, Kazuhiro Ogata, and Haruki Nakamura(*責任著者)
Nucleic Acids Research, Jan 4, 2018,
doi:10.1093/nar/gkx1297
本研究は、日本学術振興会の新学術領域、若手研究(B)、挑戦的萌芽研究、基盤研究(B)、文部科学省イノベーションシステム整備事業、および国立研究開発法人日本医療研究開発機構の支援により行われました。
参考URL
大阪大学 蛋白質研究所 附属蛋白質解析先端研究センター
http://www.protein.osaka-u.ac.jp/rcsfp/pi/index_pi.html
用語説明
- 翻訳後修飾
翻訳(タンパク質の生合成)後のタンパク質の化学修飾を指します。タンパク質は翻訳後、リン酸基、アセチル基、メチル基などの官能基が酵素的に付加されることで機能が変化し、あたかもスイッチのように機能が調節されることが知られています。翻訳後修飾は主に天然変性領域にみられます。
