全身性エリテマトーデス患者の腸内微生物叢の全体像が明らかに
メタゲノムショットガンシーケンスによる腸内微生物叢の網羅的解析
研究成果のポイント
- 腸内微生物叢由来のゲノム(メタゲノム)に対して次世代シーケンサーによるショットガンシーケンスを行い、微生物情報と全身性エリテマトーデスとの関連を網羅的に探索・同定した。
- 全身性エリテマトーデス患者の腸内微生物叢においてStreptococcus属に含まれる2種の細菌が増えていることが明らかになった。
- メタゲノム解析の結果を血中代謝物情報と統合し、腸内細菌と血中代謝物濃度の関連を明らかにした。
概要
大阪大学大学院医学系研究科の大学院生の友藤嘉彦さん(遺伝統計学)、前田悠一 助教(呼吸器・免疫内科学)、大学院生の猪頭英里さん(呼吸器・免疫内科学)、岡田随象 教授(遺伝統計学)らの研究グループは、腸内微生物叢由来のゲノム(メタゲノム)に対して次世代シーケンサーによるショットガンシーケンスを行い、微生物情報と全身性エリテマトーデスとの関連を網羅的に探索・同定しました。
我々の腸内には数多くの微生物が存在し、腸内微生物叢を構成しています。腸内微生物叢は免疫反応や代謝応答に密接に関わっており、自己免疫疾患や代謝疾患の発症に寄与することが知られています。しかし、自己免疫疾患である全身性エリテマトーデスと腸内微生物叢の関連については明らかになっていませんでした。
今回、研究グループは、全身性エリテマトーデス患者と健常者の腸内微生物叢由来のゲノム(メタゲノム)に対して、次世代シーケンサーによるメタゲノムショットガンシーケンスを行いました。そして、得られた大規模なゲノム配列情報を用いて、菌種・遺伝子・パスウェイなどの腸内微生物情報と全身性エリテマトーデスとの関連を網羅的に探索する、メタゲノムワイド関連解析を実施しました(図1)。その結果、全身性エリテマトーデス患者の腸内微生物叢において、Streptococcus属に属する2種の細菌が増加していることがわかりました。また、メタゲノムデータと質量分析計で取得した血中代謝物のデータを統合することによって、腸内微生物叢と血中代謝物との関連を見出しました。
本研究成果によって、腸内微生物叢を介した全身性エリテマトーデスの発症機序についての理解が進み、新たな治療・診断技術の開発につながると期待されます。
本研究成果は、英国科学誌「Annals of the Rheumatic Diseases」に8月25日(水)午前0時(日本時間)に公開されました。
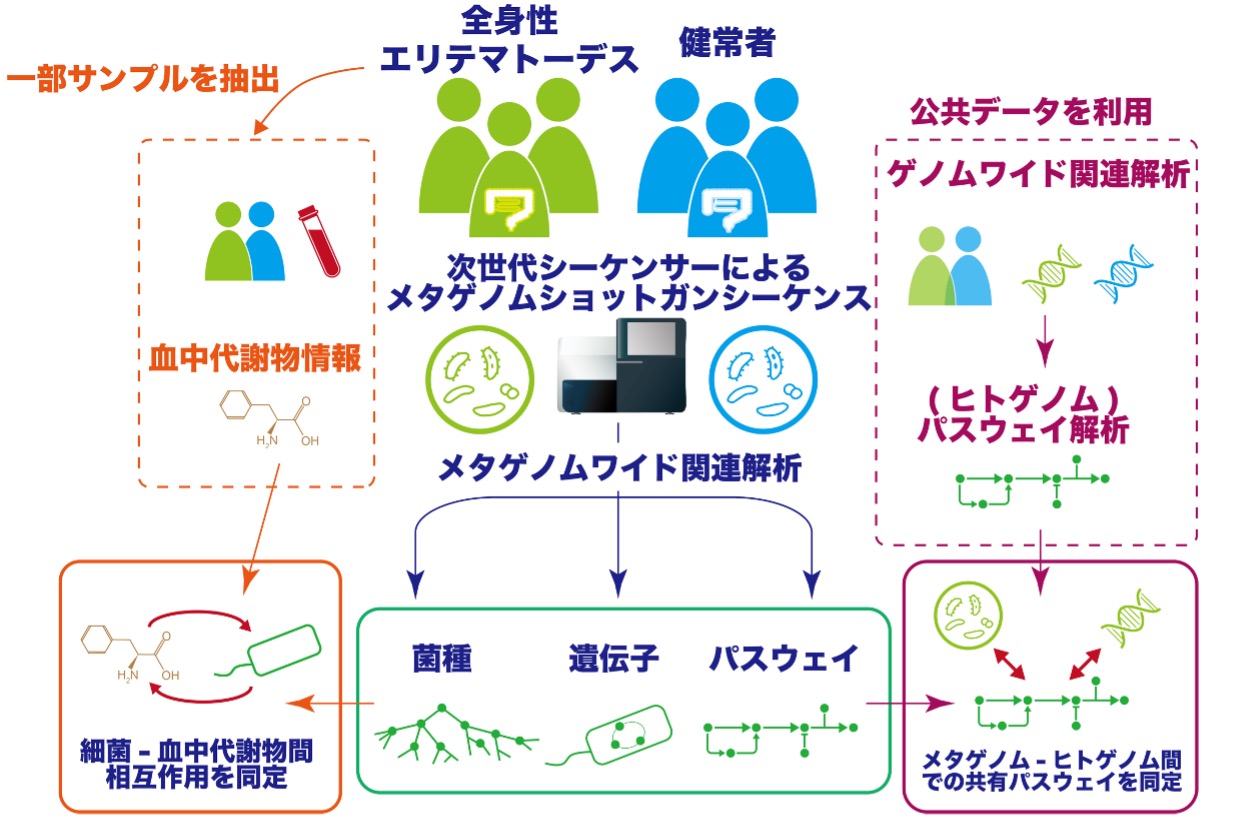
図1. 本研究の概要
研究の背景
全身性エリテマトーデスは免疫系が自己を攻撃してしまうことによって発症する自己免疫疾患の1つであり、日本にはおよそ6万人から10万人の患者がいると推定されています。全身性エリテマトーデスの発症には環境因子と遺伝因子の双方が寄与すると考えられていますが、発症機序の全容は明らかになっていません。
我々の腸内には数多くの微生物が存在し、腸内微生物叢を構成しています。腸内微生物叢は免疫反応や代謝応答を介して我々の体に大きな影響を与えており、関節リウマチをはじめとした多くの疾患との関連が既に示されています。腸内微生物叢と免疫系に密接な関係があることから、腸内微生物叢は全身性エリテマトーデスの発症に寄与する環境因子の1つではないかと考えられてきました。しかし、これまでの研究では、腸内微生物叢のうちの一部しか解析することができておらず、全身性エリテマトーデスと腸内微生物叢の関連について、その全体像を把握することができていませんでした。また、殆どの研究が日本国外で実施されたものであり、日本人集団に適用可能かどうかは不明でした。
メタゲノムショットガンシーケンスを利用したメタゲノムワイド関連解析は疾患と腸内微生物叢との関連を網羅的に探索する手法です。腸内微生物叢から得られる全てのゲノム情報(メタゲノム)を次世代シーケンサーによってシーケンスすることによって、網羅的な解析が可能になるほか、菌種、遺伝子、パスウェイといった様々な情報を得られることが特徴です。一方で、メタゲノムワイド関連解析を行うためには、膨大なメタゲノム情報を処理するために多くの計算時間が必要であり、複雑な解析を行うためのバイオインフォマティクス技術も必要となります。そのため、メタゲノムワイド関連解析は非常に有用な解析手法であるのにもかかわらず、現状ではあまり広く普及していません。
本研究の成果
今回、研究グループは、日本人集団(全身性エリテマトーデス患者47名、健常者 203名)の腸内微生物叢由来ゲノム(メタゲノム)に対して、次世代シーケンサーによるメタゲノムショットガンシーケンスを行いました。そして、得られた大規模なゲノム配列情報に対して、遺伝統計学教室で独自に開発された、菌種・遺伝子・パスウェイなどの微生物情報を網羅的に取得するパイプラインを適用しました。そして、得られた微生物情報と全身性エリテマトーデスとの関連を網羅的に探索する、メタゲノムワイド関連解析を実施しました。その結果、全身性エリテマトーデス患者の腸内微生物叢において、Streptococcus属に属する2種の細菌(Streptococcus anginosus and Streptococcus intermedius)が増加していることがわかりました(図2)。これらの菌種が全身性エリテマトーデスの病態に関与している可能性が考えられます。
また、全身性エリテマトーデス患者の腸内微生物叢では菌の多様性が低下しており、腸内微生物叢の破綻(ディスバイオシス)が起きていることも示されました。これまでに、炎症性腸疾患などの多くの疾患にディスバイオシスが関与していることが示されてきましたが、本研究によって、全身性エリテマトーデスの病態にもディスバイオシスが寄与している可能性が示唆されました。
さらに、全身性エリテマトーデス患者と健常者の間とで、腸内微生物叢由来の遺伝子・パスウェイについて比較を行ったところ、全身性エリテマトーデス患者では酸化還元反応に関与する遺伝子が増加していて、かつ硫黄代謝や鞭毛の形成に関与するパスウェイが変動していることがわかりました。これらの遺伝子・パスウェイを介した腸内微生物叢と免疫系との関連が、全身性エリテマトーデスの病態に関与している可能性が考えられます。また、パスウェイ解析の結果をゲノムワイド関連解析の結果と統合し、解析を行ったところ、メタゲノムとヒトゲノムとの間で、全身性エリテマトーデスに関与するパスウェイが共有されていることが明らかになりました。
腸内微生物叢はしばしば、我々の血中代謝物の濃度に大きな影響を与えることが知られています。そこで、メタゲノム解析の結果と質量分析計で取得した血中代謝物情報とを統合することによって、腸内微生物叢と血中代謝物との関連を探索しました。その結果、Streptococcus intermediusとアシルカルニチンとの間に存在する正の相関が見出されました。この結果から、全身性エリテマトーデス患者で増加していたStreptococcus intermediusがアシルカルニチンを介して、異常な免疫反応の活性化に寄与している可能性が示唆されました。
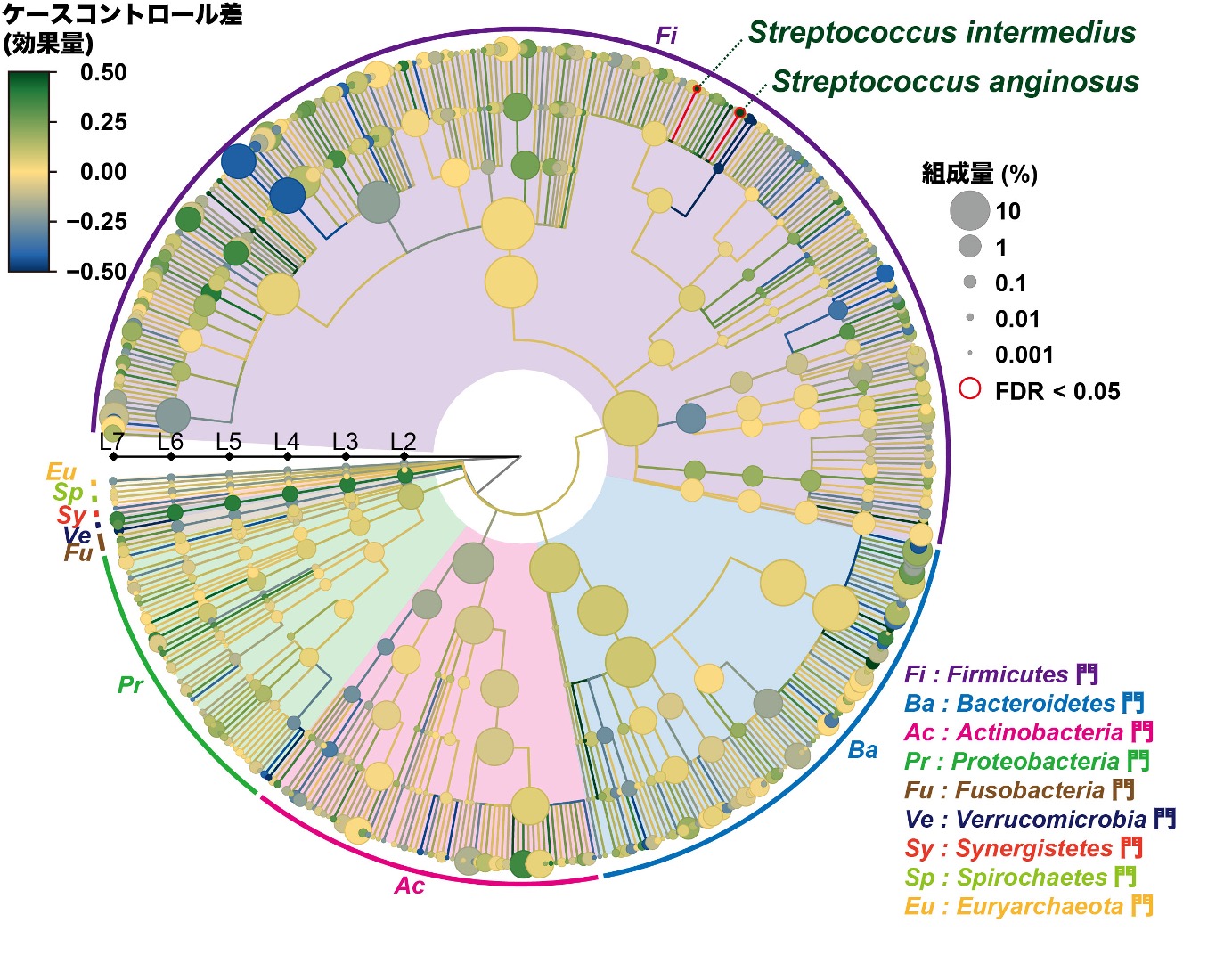
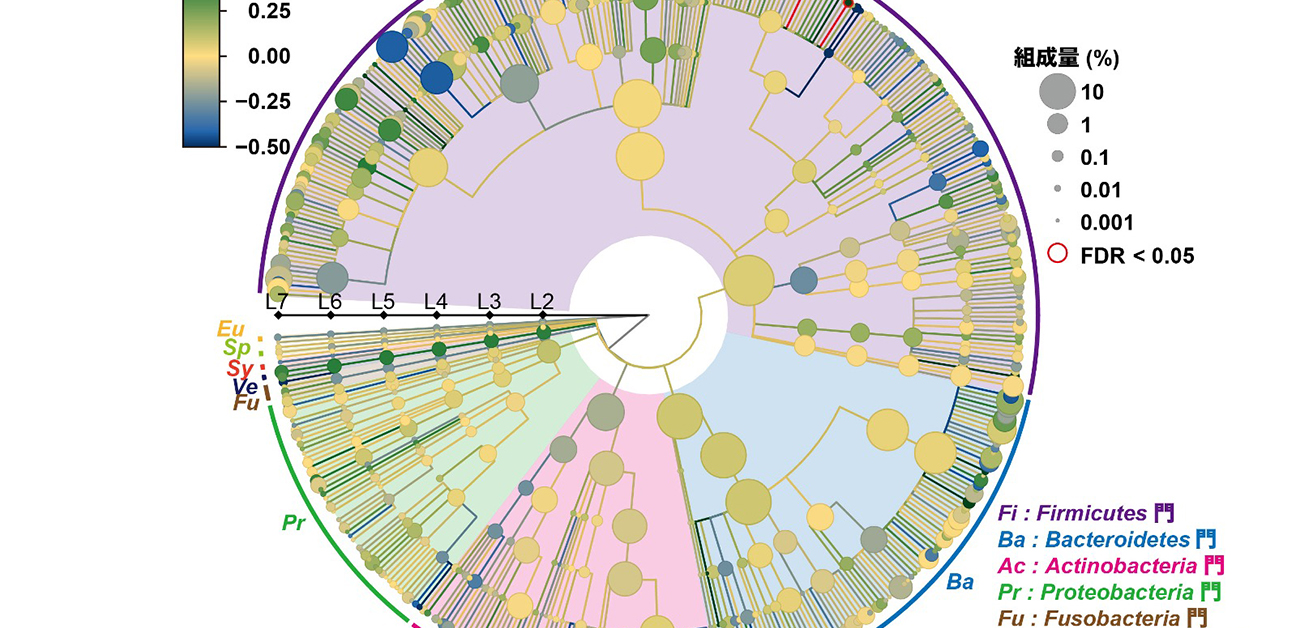
図2. 全身性エリテマトーデス患者においてStreptococcus属に属する2種類の細菌が増加 細菌の系統樹を示す。系統樹中のノードの色は、ケースコントロール解析における細菌と全身性エリテマトーデスの関連の強さ(効果量)を表している。正の効果量は正の関連を、負の効果量は負の関連を意味する。系統樹中のノードの大きさは、コホート全体における細菌の相対的存在量(組成量)の平均を表している。第一種の過誤(本来は関連がない細菌を関連があると報告してしまうこと)を抑えるために、偽発見率(False discovery ratio; FDR)を計算し、FDR < 0.05を満たす細菌を、全身性エリテマトーデスと関連のある細菌として報告した。
本研究成果が社会に与える影響(本研究成果の意義)
本研究成果によって、全身性エリテマトーデス患者における腸内微生物叢の全体像が明らかになりました。また、メタゲノム解析の結果と血中代謝物情報との統合解析によって、メタゲノム関連解析で同定されたStreptococcus intermediusの量が血中アシルカルニチン濃度と正の相関関係にあることが明らかになりました。
本研究で同定された菌種・遺伝子・血中代謝物については培養実験や動物実験などのさらなる検証を進めることで、全身性エリテマトーデスにおける治療標的としての可能性を評価することが可能であると考えられます。また、今回明らかになった、全身性エリテマトーデス患者の腸内微生物叢の特徴は、診断に利用可能なバイオマーカーとして利用可能であることが期待されます。
特記事項
本研究成果は、2021年8月25日(水)午前0時(日本時間)に英国科学誌「Annals of the Rheumatic Diseases」(オンライン)に掲載されました。
【タイトル】 “A metagenome-wide association study revealed disease-specific landscape of the gut microbiome of systemic lupus erythematosus in Japanese.”
【著者名】Yoshihiko Tomofuji1,13, Yuichi Maeda2,3,4,13, Eri Oguro-Igashira2,3,13, Toshihiro Kishikawa1,5,6, Kenichi Yamamoto1,7, Kyuto Sonehara1,4, Daisuke Motooka8, Yuki Matsumoto8, Hidetoshi Matsuoka9, Maiko Yoshimura9, Mayu Yagita2,3, Takuro Nii2,3, Shiro Ohshima9, Shota Nakamura4,8, Hidenori Inohara5, Kiyoshi Takeda3,10, Atsushi Kumanogoh2,4,11, Yukinori Okada1,4,12
【所属】
1. 大阪大学大学院医学系研究科 遺伝統計学
2. 大阪大学大学院医学系研究科 呼吸器・免疫内科学
3. 大阪大学大学院医学系研究科 免疫制御学
4. 大阪大学先導的学際研究機構 生命医科学融合フロンティア研究部門
5. 大阪大学大学院医学系研究科 耳鼻咽喉科・頭頸部外科学
6. 愛知県がんセンター 頭頸部外科部
7. 大阪大学大学院医学系研究科 小児科学
8. 大阪大学 微生物病研究所 感染症メタゲノム研究分野
9. 大阪南医療センター リウマチ・膠原病・ アレルギー科
10. 大阪大学 免疫学フロンティア研究センター 粘膜免疫学
11. 大阪大学 免疫学フロンティア研究センター 感染病態分野
12. 大阪大学 免疫学フロンティア研究センター 免疫統計学
13. 共同筆頭著者
【DOI番号】10.1136/annrheumdis-2021-220687
本研究は、日本医療研究開発機構(AMED)革新的先端研究開発支援事業(FORCE)の採択課題「メタゲノムワイド関連解析による疾患特異的微生物叢解明と個別化医療実装」(研究開発代表者:岡田随象)の一環として行われ、大阪大学免疫学フロンティア研究センター 次世代主任研究者支援プログラム、大阪大学先導的学際研究機構、大阪大学大学院医学系研究科 バイオインフォマティクスイニシアティブの協力を得て行われました。
用語説明
- 腸内微生物叢
宿主であるヒトや動物と共生関係にある多種多様な腸内微生物の集まり。
- メタゲノム
群集を構成する微生物のゲノムの総和。各微生物の単離・培養を行わず、集団から直接収集・抽出される。
- 次世代シーケンサー
数千から数百万ものDNA分子を同時に配列決定するシーケンシング機器。
- ショットガンシーケンス
微生物の全ゲノムDNAを短いDNA鎖に切断してライブラリを作成し、次世代シーケンサーによって配列決定する手法。
- パスウェイ
遺伝子やタンパク質の相互作用、細胞内の連鎖的な化学反応など、生体内における様々なネットワークを示す経路。
- 質量分析計
物質の質量を測定する装置。血中代謝物の種類や量についての情報を取得することができる。
- 関節リウマチ
自己免疫疾患の1つで、関節に炎症が起き、骨が破壊されて、放っておくと関節が変形してしまう病気。
- バイオインフォマティクス
生命科学と情報科学の融合分野のひとつであり、情報科学や統計学などのアルゴリズムを用いた方法論やソフトウェアを活用して生命現象を解き明かすことを目的とする。
- 炎症性腸疾患
自己免疫疾患の1つで、自分の免疫細胞が腸の細胞を攻撃してしまうことで腸に炎症を起こす病気。
- 鞭毛
ある種の細菌などにみられる、細長い突起状をした運動性の細胞小器官。免疫応答を引き起こすことが報告されている。
- ゲノムワイド関連解析
ヒトゲノム配列上に存在する数百万カ所の遺伝子変異を網羅的にタイピングし、疾患や形質との関連を網羅的に探索する手法。
- アシルカルニチン
脂肪酸とカルニチンが結合して生成する化合物。炎症との関連が報告されている。
- バイオマーカー
タンパク質や遺伝子などの生体内の物質で、病状の変化や治療の効果の指標となるもの。
