見えた!ゲノムDNAを修復する酵素のすがた
高速原子間力顕微鏡により解明された分子レベルの酵素の動き
研究成果のポイント
・放射線から私たちのゲノムDNAを守るヒトDNA修復酵素MRE11/RAD50/NBS1(MRN)の分子レベルの構造や動きを解明した。
・明らかとなったMRNの分子構造は欧米のトップ研究グループにより長年提唱されてきた定説を覆すものであり、MRNが働く仕組みの理解を大きく深めるものであった。
・ヒトMRNはゲノムDNAを修復する酵素だがゲノム編集技術にも重要な酵素であり、今回の発見の医学・生物学的意義は大きい。
概要
大阪大学蛋白質研究所の古郡麻子准教授らは名古屋大学大学院理学研究科内橋貴之教授、奈良先端科学技術大学院大学建部恒助教らとの共同研究によりヒトのゲノムDNAを修復する酵素であるMRE11/RAD50/NBS1複合体(MRN)の高速原子間力顕微鏡観察に成功しました。
放射線や抗がん剤などによってゲノムDNAが切断されてしまうとヒトの細胞は死んでしまいます。この切れたゲノムを治すために細胞の中で働いている酵素がヒトMRNです。そのためMRNは抗がん剤や放射線治療に関係するほか、細胞のMRNの働きが悪くなると発がんや老化が促進されます。またゲノム編集技術でもゲノムDNAの切断を人為的に誘導するため細胞のMRNが働かなければゲノム編集はできません。
このように医学・生物学的に重要な酵素であるMRNの分子構造を明らかにするため、主に欧米の研究グループにより原子間力顕微鏡による蛋白質構造解析が20年ほど前から精力的に行われてきました。しかし今回、日本で開発された高性能の高速原子間力顕微鏡によるMRNの分子構造観察を行ったところ、これまで報告されていた酵素の姿とは全く異なる像が見えてきました( 図1 、 図2 )。
これまでMRNは主に球体から二本の腕を伸ばしたような形をしていると報告されていて (図2右端) 伸ばした腕はMRNが放射線により切断されたDNAを修復する際にDNAを繋ぐために使われると考えられてきました。しかし今回、古郡准教授、内橋教授によって実施された高速原子間力顕微鏡観察により、MRNは主にはリング状の構造をしており、球体部分が二つに別れてサクランボ状の構造になることでリング構造の開閉をしていることが明らかになりました( 図2、 図3 )。また、今回観察された分子構造の正しさは、建部助教による分裂酵母を用いた遺伝学解析によって生物学的にも証明されました。
本研究成果は、英国科学誌「Nature Communications」に、1月17日(金)19時(日本時間)に公開されました。

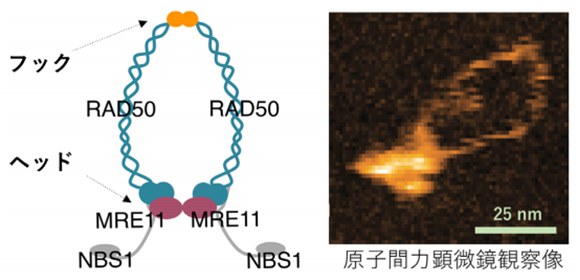
図1 ヒトMRN複合体の分子構造
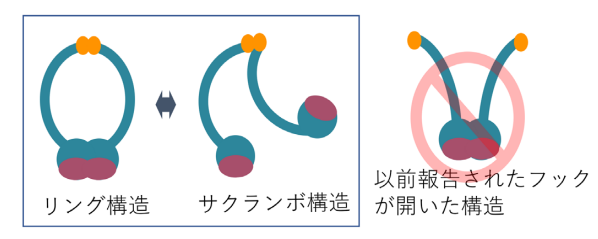
図2 観察されたヒトMRN分子の動き(模式図)
研究の背景
細胞のゲノムDNAは遺伝情報を含んでいて、その塩基配列は細胞の働きを決める生命の設計図とも言えるものです。このゲノムDNAが放射線や発がん物質などによって傷つけられると、遺伝情報が失われ細胞が癌化したり死んでしまったりします。それを防ぐために、傷ついたゲノムDNAを修復するための仕組みが細胞には備わっています。本研究で対象としているヒトMRNは、特に放射線や活性酸素、発がん物質などによって引き起こされるゲノムDNAの切断を修復するメカニズムで重要な働きを担っている酵素です。例えば放射線治療では正常な細胞は少々ゲノムDNAが傷ついても死にませんが、MRNの働きが弱くなっている細胞はゲノムDNAの傷を治せずに死んでしまいます。また特に放射線などを当てなくてもDNAの切断は低頻度ながら起きるため、MRNは私たちが生きていく上で欠くことのできない必須の酵素です。
本研究の内容(本研究成果の意義)
このように重要な酵素にもかかわらず、MRNがどのように切れてしまったDNAを直しているのかについては、実はよくわかっていませんでした。その中でも大きな謎の一つとされてきたのがMRN蛋白質分子の不思議なリング状の形状です。MRNはMRE11ヌクレアーゼ、RAD50、NBS1の三種類の蛋白質が各2個、全部で6個の蛋白質がリング構造を形成した複合体です (図1) 。
長年、このリング構造がフックと呼ばれる部位 (図1) 、模式図のオレンジ色の部位)で開くことがDNAの傷を直す上で重要とされてきましたが、本研究では定説とは逆側のヘッドと呼ばれる球状ドメイン 側で開き、サクランボ構造となることがわかりました( 図2左 、 図3 )。さらに本研究ではヒトのみならず酵母や大腸菌のMRNホモログ の分子構造を調べ、新たに発見されたヘッド部位でのリング開閉運動が種を超えて保存されていることも見つけました。このことから、この動きがMRNの本質的な性質であることを示していると考えられます。さらにMRNのリング構造がフックでは開かないことを証明するため、生きた酵母細胞中のMRNのフックをSMCタンパク質 と呼ばれる蛋白質のヒンジドメインと入れ替えたキメラMRN を持つ酵母がDNAの傷を治す力があることを示しました。このようにMRNはフックではなくヘッドでリングを開閉することが明らかになったことから、MRNはDNAを修復する際にリングの内側にDNAを包み込む形で結合する可能性が示されました。
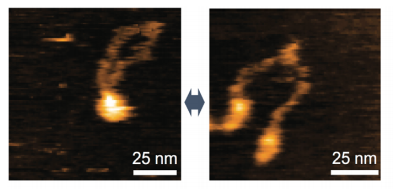
図3 観察されたMRNー分子の映像
高速原子間力顕微鏡観察により,MRNがリング構造からサクランボ構造へと変化する様子を捉えた。
本研究成果が社会に与える影響(本研究成果の意義)
蛋白質1分子のサイズはナノメートル単位と非常に小さく、通常の顕微鏡では観察することができません。そのため蛋白質分子の形状や溶液中の動きを調べるために、観察対象とプローブの間の原子間力を測定することで可視化する『原子間力顕微鏡』と呼ばれる特殊な顕微鏡が使われます。本研究で使用された『高速原子間力顕微鏡』は金沢大学を中心に開発されたもので、蛋白質1分子の溶液中での動きを1フレームあたり最速100ミリセカンドの動画で撮影できる画期的な装置です。これを用いてヒトMRNの溶液中の動きを調べることで上記の発見を成し遂げることができました。
本研究でMRNの分子の形がわかったからといって一足飛びにMRNの働きの全てが解明されるわけではありませんし、すぐさま医療に応用できるわけではありません。しかし、酵素の形や動きの理解はその酵素の働きや制御方法の理解の第一歩となる重要な基礎研究です。こうした基礎研究をもとに、将来、もし細胞中のMRNを効果的に制御することが可能になれば、放射線治療や抗がん剤の改良や新たなゲノム編集技術の開発などの応用研究に役立つことが期待されます。
特記事項
本研究成果は、2020年1月17日(金)19時(日本時間)に英国科学誌「Nature Communications」(オンライン)に掲載されました。
タイトル:“Rad50 zinc hook functions as a constitutive dimerization module interchangeable with SMC hinge”
著者名:Hisashi Tatebe,Chew Theng Lim, Hiroki Konno, Kazuhiro Shiozaki, Akira Shinohara,Takayuki Uchihashi and Asako Furukohri
(建部恒、Lim Chew Theng、紺野宏記、塩﨑一裕、篠原彰、内橋貴之、古郡麻子)
なお、本研究は、科学研究費補助金研究(17H05883,19H03157,18H01837,16K07255,18H04512,19H05389,19K06542)および蛋白質研究所新分野開拓支援プロジェクトの一環として行われました。
参考URL
大阪大学 蛋白質研究所 ゲノムー染色体機能研究室
http://www.protein.osaka-u.ac.jp/genome/
用語説明
- ドメイン
一つの蛋白質は均一な球状をしているのではなく、独立した構造を持ついくつかの機能単位を形成していることが多く、この機能単位をドメインと呼ぶ。MRNのヘッドドメインは腕部分とは異なり球状の形をしており、ATPアーゼ活性とヌクレアーゼ活性の酵素活性を持つ。
- ホモログ
異なる生物種には非常によく似た蛋白質が存在することがある。これらは共通の祖先を持ち、進化の過程で少しずつ変化するものの多くの場合性質や役割は保存されていることが多い。生物の共通祖先がすでにMRNを持っていたため、現存の生物は全てMRNのホモログを持つと考えられている。
- SMCタンパク質
染色体の構造を制御する蛋白質群で、MRNによく似たリング構造を持つ。フックの代わりにヒンジと呼ばれるドメインでリング構造を閉じていることが知られている。
- キメラMRN
一つの蛋白質分子の中に起源の異なる部位が混じり合った蛋白質をキメラ蛋白質と呼ぶ。本研究ではMRNの中にSMCのヒンジを持たせたキメラMRNを作り出し、それが分裂酵母中で本来のMRNに代わって働き得るかを調べた。
