\階層的分類システム×機械学習/ 新技術scODINにより 細胞の高速・高精度解析を実現
研究成果のポイント
概要
大阪大学感染症総合教育研究拠点のJonas Søndergaard招へい研究員、James Badger Wing教授らの研究グループは、単一細胞RNAシーケンス解析において、新たな解析手法「scODIN」を開発しました。
「scODIN」は、階層的分類システムと機械学習を融合した手法で、single-cell RNA-seq データにおける名前の最適化された検出と推論(optimized detection and inference of names in scRNA-seq data)を可能にします。
これにより、これまで見逃されていた希少細胞や移行期細胞を高速・高精度で自動分類できることを世界で初めて明らかにしました。本手法は、数十万個の細胞を含む大規模データをわずか数分で処理でき、従来の手法では困難だった細胞状態の連続的な変化を正確に捉えることが可能です。
これまで単一細胞RNAシーケンス解析では、処理時間と分類精度がトレードオフの関係にあると考えられており、希少細胞や中間状態の細胞の同定については解明されていませんでした。今回、研究グループは独自の階層的分類システム(Tier system)と機械学習(k近傍法)を組み合わせることにより、65万個の細胞をわずか6分で処理しながら、従来法を大幅に上回る分類精度を達成できることを解明しました。この成果により、疾患の病態解明や創薬標的の探索、個別化医療の実現が期待されます。
本研究成果は、2025年8月21日に米国科学誌「The Journal of Immunology」(オンライン)に掲載されました。
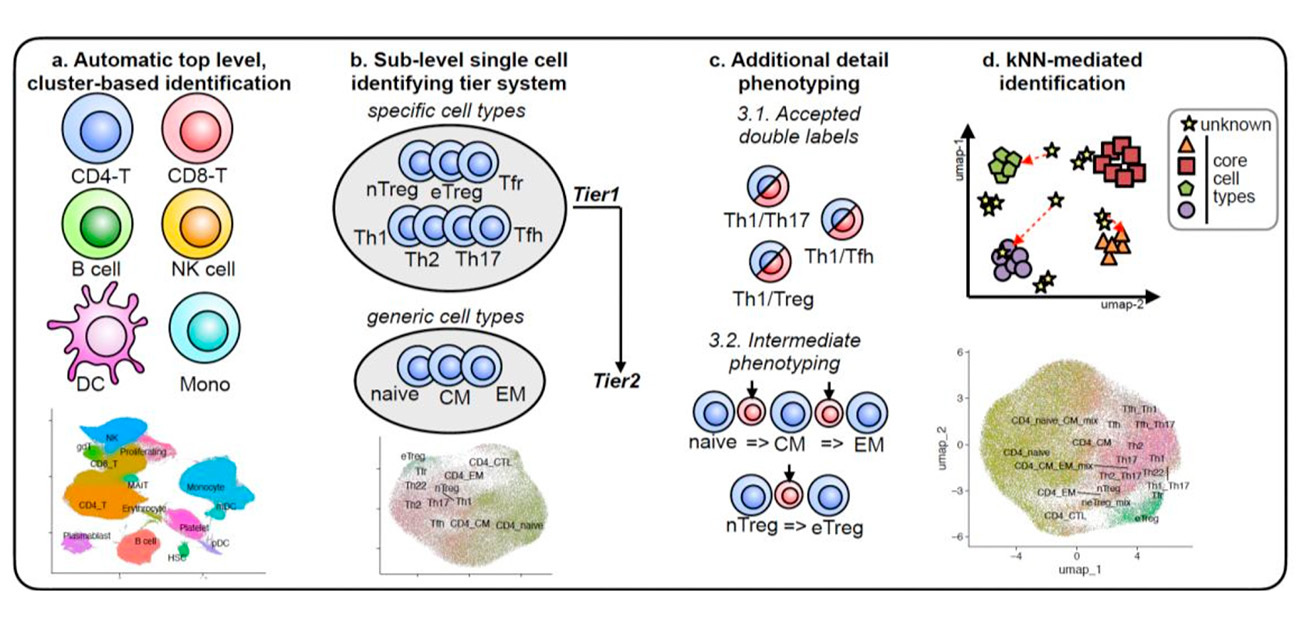
図1. scODIN (Optimized Detection and Inference of Names in scRNA-seq data) の概要
a, 主要な細胞クラスター(CD4 T細胞、B細胞、単球)を自動で高速かつ正確に同定し、さらなる詳細な分類を行う。
b, 階層的分類システムにより、詳細なレベルで細胞を同定(Tier 1で制御性T細胞(Treg)やヘルパーT細胞(Th細胞)などのサブセットを識別し、Tier 2でセントラルメモリー(CM)やエフェクターメモリー(EM)などの細胞型を識別)。
c, さらに二重標識ラベルを追加することで、移行期細胞を同定。
d, 最後のステップではk近傍法(kNN)推論を用いることで、すべての細胞をカテゴリー化されます。
研究の背景
これまで、単一細胞RNAシーケンス解析は、個々の細胞の遺伝子発現を網羅的に測定できる強力な技術であることが知られていました。しかし、データが大規模化するにつれ、細胞の自動分類において処理速度と分類精度のトレードオフ、希少細胞の見逃し、移行期細胞の誤分類などの課題がありました。特に免疫細胞は多様な亜集団を持ち、疾患により微妙に変化するため、詳細かつ正確な分類が求められていました。
研究の内容
研究グループでは、階層的分類システム(Tier system)と機械学習(k近傍法)を組み合わせた独自の方法により、大規模な単一細胞RNAシーケンスデータでも高速・高精度な細胞分類を可能にする「scODIN」を開発しました。これは、まず主要な細胞タイプを分類(Tier1)した後、さらに詳細な亜集団を同定(Tier2)する二段階アプローチで、従来法では困難だった希少細胞(Th22細胞など)や移行期細胞(ナイーブ→メモリー移行中など)の正確な同定を実現しました。
特筆すべき点として、本手法は約65万細胞を含む大規模データセットをわずか6分で処理でき、従来法で数時間を要していた処理時間を劇的に短縮しました。
さらに、COVID-19患者データへの適用により、疾患特異的な免疫細胞の変化を詳細に解析することを実現し、臨床応用への可能性を示しました。
本研究成果が社会に与える影響(本研究成果の意義)
本研究成果により、あらゆる疾患の免疫応答を詳細に解析することが可能となり、病態メカニズムの解明や新規治療標的の発見が期待されます。特に、がん免疫療法の効果予測、自己免疫疾患の早期診断、感染症における重症化予測など、個別化医療の実現に貢献することが期待されます。また、本手法はオープンソースとして公開予定であり、世界中の研究者が利用可能となることで、免疫学研究全体の加速に寄与すると考えられます。
特記事項
本研究成果は、2025年8月21日に米国科学誌「The Journal of Immunology」(オンライン)に掲載されました。
タイトル:“Optimized Detection and Inference of immune cell type Names in scRNA-seq data”
著者名:Janyerkye Tulyeu, David Priest, James B. Wing*, Jonas Nørskov Søndergaard*
DOI:https://doi.org/10.1093/jimmun/vkaf183
参考URL
James Badger Wing教授 研究者総覧
https://rd.iai.osaka-u.ac.jp/ja/0d19f8bd3f186a96.html
感染症総合教育研究拠点(CiDER) 研究者紹介
https://www.cider.osaka-u.ac.jp/researchers/james-wing/
SDGsの目標
用語説明
- 単一細胞RNAシーケンス解析
個々の細胞から抽出したRNA(遺伝子の発現情報)を網羅的に測定する技術。従来の解析では細胞集団の平均値しか得られなかったが、この技術により一つ一つの細胞の特徴を詳細に調べることが可能。
- 階層的分類システム
Tier system。scODINで採用された段階的な細胞分類手法。第1階層(Tier1)で主要な細胞タイプ(T細胞、B細胞など)を分類し、第2階層(Tier2)でより詳細な亜集団(Th1、Th2、Th17など)を同定する。
- 希少細胞
全体の細胞集団の中で非常に少ない割合(1%未満など)でしか存在しない細胞。Th22細胞やTfr細胞など、重要な機能を持つにも関わらず、従来の解析手法では見逃されることが多かった。
- 移行期細胞
ある細胞状態から別の細胞状態へ変化している途中の細胞。例えば、ナイーブT細胞からメモリーT細胞への分化過程にある細胞など。連続的な変化の中間状態を捉えることで、細胞の運命決定メカニズムの理解につながる。

