マウス・ラット等の遺伝子改変効率を向上させる新しい技術を開発
従来の約100倍のサイズのゲノム編集が可能に!
本研究成果のポイント
・ゲノム編集技術‘CRISPR/Cas システム’ と一本鎖オリゴ(ssODN) を利用する二つの新しい遺伝子改変技術の方法を開発
・これにより、動物の受精卵において、これまで難しかった数百kbの大きさの染色体領域のゲノム導入に成功
・マウスやラットなど様々な動物における遺伝子改変操作の効率を向上させるとともに、作製された遺伝子改変動物 は、創薬研究、トランスレーショナル研究、再生医療研究などへの幅広い利用に期待
概要
大阪大学大学院医学系研究科附属動物実験施設の真下知士(ましも ともじ)准教授、情報・システム研究機構国立遺伝学研究所マウス開発研究室の吉見一人(よしみ かずと)助教らの研究グループは、ゲノム編集技術‘CRISPR/Cas システム’と一本鎖オリゴ(ssODN)を利用する二つの新しい遺伝子改変技術の方法(「lsODN(長鎖一本鎖DNA)法」 と「2H2OP(2ヒット2オリゴ)法」 (図1) )を開発しました。
‘CRISPR/Casシステム’は、マウスやラットにおける新しい遺伝子改変技術として注目されている技術です。DNAを切断する酵素Cas9と、ゲノム上の編集箇所を見つけ出すgRNAを動物の受精卵に注入することで、特定の遺伝子を破壊(ノックアウト)したり、特定の箇所へ導入(ノックイン)することができます。しかしながらこれまで動物の受精卵では、遺伝子などの大きなDNA配列の導入効率が低く、ノックイン動物を作製することが困難でした。
本研究で開発した二つの新しい遺伝子改変技術の方法により、GFP遺伝子 の効率的かつ正確なノックインに加え、これまで不可能だった大きなサイズのゲノム領域(約200 kbp)の導入、ラット遺伝子のヒト由来遺伝子への置き換え(遺伝子ヒト化動物)に成功しました (図2) 。
今後、これら二つのノックイン法は、マウスやラットなどのみならず様々な生物種における遺伝子改変操作の効率を向上させ、新しい遺伝子組み換え生物の作製に非常に有用な技術になることが期待されます。また、作製された遺伝子改変動物は、創薬研究、トランスレーショナル研究、再生医療研究などへの幅広い利用が期待されます。
本研究成果は、平成28年1月20日(水)に英国ネイチャー出版グループ オープンアクセス誌「Nature Communications」から公開されました。
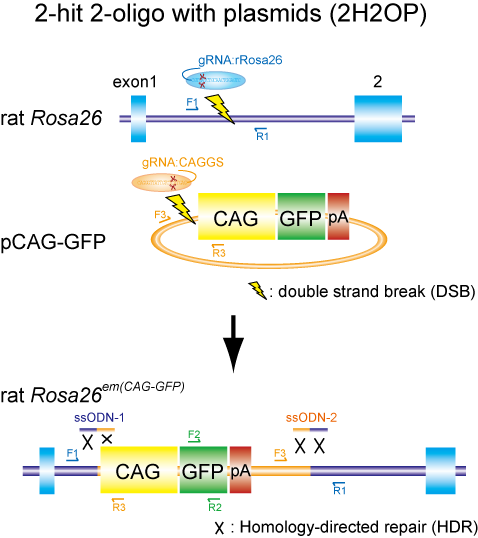
図1 2H2OP法の略図
一本鎖DNA(ssODN)を‘のり’として利用することで、プラスミドを標的部位へ効率的にノックインできる。

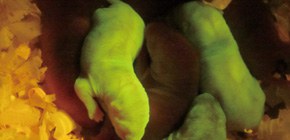
図2 2H2OP法により作製されたGFPノックインラット。
ノックイン個体は全身で緑色蛍光タンパク質を発現するため、全身が緑色の蛍光を発する。
研究の背景と成果
CRISPR/Casの登場により、マウス、ラットなどにおける遺伝子改変が簡単になりました。動物の受精卵にCas9のメッセンジャーRNAとgRNAを導入することで、gRNAが標的となるDNA配列を認識し、Cas9がその標的部位を切断します。切断されたDNA部位は非相同末端結合(non-homologous end joining)により修復されますが、その際に、DNA変異が導入されることで、遺伝子を破壊(ノックアウト)できます。また、Cas9/gRNAと一緒に、ドナーDNAとなる一本鎖オリゴ(ssODN)を導入すると、ドナーDNAを利用した相同性修復(homology-directed repair)により切断部位が修復され、1~数十塩基(bp)のDNA配列を導入(ノックイン)することができます。しかしながら、これまでssODNは200bp程度までしか合成できず、そのためssODNを用いてGFPなどの大きなサイズのDNA配列をノックインすることが困難でした。一方、より長い配列を保持できるDNAプラスミドをドナーDNAとして用いた場合、受精卵では相同組換え効率が低いため、プラスミドDNAをノックインすることが困難でした。
本研究の成果は以下になります。
1) Cas9発現プラスミドのターミネーター配列にポリアデニン鎖polyAを付加することで、受精卵でのCas9タンパク質の発現が上昇しました。このCas9-polyAを利用して、受精卵でのゲノム編集効率を向上させることができました。
2) バイオダイナミクス研究所との共同研究により、長鎖の一本鎖DNA(lsODN)の作製方法を新たに開発し、lsODNをドナーDNAとして用いることで効率的および正確にGFP配列をノックインすることに成功しました。
3) CRISPR/Casシステム(Cas9 mRNA、二つのgRNA)を‘はさみ’として、一本鎖DNA(ssODN)を‘のり’として利用する方法(2ヒット2オリゴ法:2H2OPと命名)を開発し、CAG-GFP配列5.5 kbpのプラスミドを標的部位へ効率的にノックインしました。
4) この2H2OP法を利用することで、これまでノックインが不可能だった大きなサイズ(200-kbp)であるBACプラスミド (ヒトSIRPA遺伝子配列全体を含む)をノックインすることに成功しました。
5) 最後に、三つのgRNA を利用する3H2OP法により、DNA切断部位を増やすことで、ラット Cyp2d クラスター遺伝子座58kbpをヒト CYP2D6 遺伝子6.2kbpに置換することに成功しました。
本研究成果が社会に与える影響(本研究成果の意義)
今回、CRISPR/Casシステム と一本鎖オリゴを組み合わせることで大きなサイズのDNA配列を効率的にノックインする二つの新しい方法(lsODN法と2H2OP法)を開発しました。1) lsODNを利用することで、GFPやDsRedなどのレポーター遺伝子を正確にノックインできるため、マウスやラットの遺伝子を標識することができるようになりました。2) 2H2OP法を利用することで、これまで不可能であったBACプラスミドのノックイン、遺伝子クラスターを含むDNA領域の置換などが可能になりました。例えば、マウスやラットの遺伝子をノックアウトすると同時に、ヒト遺伝子をノックインした「遺伝子ヒト化動物」 の作製が可能です。
今回の新しいノックイン技術は、マウスやラットなど様々な動物における遺伝子改変操作の効率を向上させるとともに、作製された遺伝子改変動物は、創薬研究、トランスレーショナル研究、再生医療研究などに幅広く利用されることが期待されます。
特記事項
<研究助成>
本研究の一部は、以下の事業の助成を受けて実施されました。
・独立行政法人日本学術振興会 科学研究費助成事業(基盤研究(B))
「実験用ラットにおけるゲノム編集基盤技術の開発」(課題番号:26290033、代表:真下知士)
・独立行政法人日本学術振興会 科学研究費助成事業(研究活動スタート支援)
「CRISPR/Cas9を用いた多重遺伝子ノックアウトラット作製技術の開発」(課題番号:25890011、代表:吉見一人)
<掲載論文>
‘ssODN-mediated knock-in with CRISPR-Cas for large genomic regions in zygotes’
「CRISPR-Casシステムと一本鎖オリゴによる受精卵での大きなゲノム領域のノックイン」
Kazuto Yoshimi 1,2 , Yayoi Kunihiro 1,3 , Takehito Kaneko 1 , Hitoshi Nagahora 4 , Birger Voigt 1 and Tomoji Mashimo 1,3
吉見 一人 1,2 、国広弥生 1,3 、金子武人 1 、長洞仁 4 、フォークト ビルガー 1 、真下知士 1,3
1 Institute of Laboratory Animals, Graduate School of Medicine, Kyoto University, Kyoto 606-8501,
2 Mouse Genomics Resource Laboratory, National Institute of Genetics, Shizuoka 411-8540,
3 Institute of Experimental Animal Sciences, Graduate School of Medicine, Osaka University, Osaka 565-0871,
4 BioDynamics Laboratory Inc., Tokyo 113-0033, Japan
1 京都大学大学院医学研究科附属動物実験施設
2 国立遺伝学研究所系統生物研究センターマウス開発研究室
3 大阪大学大学院医学系研究科附属動物実験施設
4 株式会社バイオダイナミクス研究所
DOI番号:10.1038/NCOMMS10431
Nature Communications: http://www.nature.com/naturecommunications
参考URL
論文掲載先(Nature Communications (DOI:10.1038/NCOMMS10431))
http://www.nature.com/ncomms/2016/160120/ncomms10431/full/ncomms10431.html
大阪大学大学院医学系研究科実験動物学 真下研究室
http://www.iexas-osaka-u.jp/lab/
研究内容(大阪大学大学院医学系研究科・医学部HPより)
http://www.med.osaka-u.ac.jp/jpn/activities/research/shisetsu.html#R02
用語説明
- CRISPR/Cas システム
CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats)とは、細菌や古細菌のゲノムDNAに存在する繰り返し配列のことで、1987 年、大阪大学中田篤男らにより初めて発見されました。CRISPRから転写されたガイドRNA(gRNA)が標的とするDNA配列を認識して、DNAを切断する酵素CRISPR-associated protein(Cas9)と一緒に複合体として、標的とするDNA配列を切断します。このCRISPR/Casシステムを利用することで、細胞や植物、動物などで効率的な遺伝子改変(ゲノム編集)が可能となりました。
- 一本鎖オリゴ
一本鎖オリゴ(ssODN: single-stranded oligodeoxynucleotide)とは、一本鎖の直鎖状DNAのことです。CRISPR/Casシステムと一緒に、通常、両末端に40-60bpの相同配列(homology arms)が付加されたssODNをドナーDNAとして利用することで、効率的なノックインが可能です。ssODNは化学合成により作られますが、安定して正確に合成できる最大長は、現時点では200塩基程度です。
- 遺伝子改変動物
遺伝子改変動物は、遺伝子の機能を個体レベルで調べる目的で、あるいは、ヒト遺伝性疾患のモデル動物として利用されています。遺伝子改変動物には、遺伝子を破壊する‘ノックアウト’と遺伝子を挿入する‘ノックイン’の2種類があります。遺伝子改変動物を作製するためには、これまで、胚性幹(ES)細胞が利用されてきましたが、CRISPR/Casシステムの登場により、短期間で効率的に行われるようになりました。
- lsODN(長鎖一本鎖DNA)法
1から3k(キロ)bpほどの長い一本鎖オリゴ(lsODN: long single-stranded oligodeoxynucleotide)のことで、バイオダイナミクス研究所との共同研究により、作製することに成功しました。作製したlsODNは、ssODNと同様にドナーDNAとして利用でき、CRISPR/Casシステムを組み合わせることで効率的にノックインできます。
- 2H2OP(2ヒット2オリゴ)法
プラスミドDNAなどの大きなDNA配列を導入するために開発した方法で、CRISPR/Casシステムを‘はさみ’として、一本鎖オリゴ(ssODN)を‘のり’として利用します。ゲノム上の標的配列と、導入したいプラスミドDNAを、それぞれ設計した二つのgRNA(はさみ)で切断し、切断点を結合させるために二つのssODN(のり)を利用することで、相同配列がなくても効率的にプラスミドを導入することができます。
- GFP遺伝子
2008年にノーベル化学賞を受賞した下村脩氏によって発見された緑色蛍光タンパク質(GFP)をコードする遺伝子。GFP遺伝子を動物へ導入すると、体内でGFPが合成され、その組織・細胞は緑色の蛍光を発します。こうしたGFPなどのレポーター遺伝子をゲノム上の標的遺伝子の部分に導入することで、その遺伝子が機能する時期や場所を明らかにすることができ、生体内における生命現象を可視化することができます。
- 遺伝子ヒト化動物
ゲノム編集技術によりヒト遺伝子をノックインすることで、ヒト遺伝子機能を持ったマウス・ラットを作製することができます。例えば、肝臓でヒト特有の代謝機能に関連するp450遺伝子群や、ヒト免疫反応に関するMHC遺伝子群などをノックインすることで作製できます。作製されたヒト化動物は、動物とは異なるヒト特有の生理的反応を示すことが期待され、薬物安全性試験や毒性試験、ヒト病態モデルなどに利用できます。
- BACプラスミド
BAC (bacterial artificial chromosome) とは、大腸菌由来の人工染色体プラスミドのことです。100~250 kbp程の長い染色体領域をDNA断片としてクローン化することができることから、ヒト全ゲノム領域をクローニングしたBACライブラリーが作製されています。
