DNA2本鎖が切断された場所に修復タンパク質が集まる仕組み
研究成果のポイント
・DNA2本鎖の切断を修復するために必要なタンパク質RNF168 がDNA損傷部位に集まる仕組みを明らかにしました。
・RNF168が複数の機能ユニットを利用してDNA損傷部位の目印となるユビキチン鎖 を認識する分子機構を明らかにしました。
・本成果は、DNA2本鎖切断の修復で重要な一過程の分子機構を明らかにしたもので、細胞のがん化や放射線感受性を抑えるための基盤となる知見になると期待されます。
概要
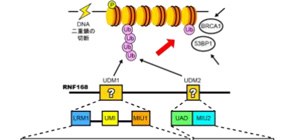
東京大学分子細胞生物学研究所(所長 白髭克彦)の深井周也准教授、大阪大学の中田慎一郎教授(高等共創研究院/大学院医学系研究科)らの研究グループは、DNA2本鎖の切断を修復するために必要なタンパク質RNF168が二つのユビキチン鎖認識ドメイン(UDM1およびUDM2)を介してユビキチン鎖を認識してDNA損傷部位に集まる仕組みを明らかにしました (図1) 。これまでの研究により、RNF168に含まれるUDM1とUDM2の二つの領域がユビキチンの認識に関与していることが示唆されていましたが、この二つの領域内に含まれるどの機能ユニットがユビキチンを直接認識しているのかは不明でした。深井准教授らの研究グループは、UDM1およびUDM2がユビキチン鎖と結合した状態の立体構造を決定し、さらに分子・細胞レベルでの変異体解析を行うことで、新たな機能ユニットを含む複数の機能ユニットがユビキチン鎖を認識する様子を明らかにしました。本研究成果は、DNA2本鎖切断の修復で重要な一過程の分子機構を明らかにしたもので、細胞のがん化や放射線感受性を抑えるための基盤となる知見になると期待されます。
研究の背景
最も重篤なDNA損傷の一つであるDNA2本鎖の切断は複数の反応過程を経て修復されます。DNAは通常、四種類のコアヒストンタンパク質が二つずつ入ったヒストン八量体に巻きついた状態で存在します。ヒストン八量体とそれに巻きついたDNAを一単位とした構造をヌクレオソームと呼び、ヌクレオソームが数珠状につらなった構造をクロマチンと呼びます。DNA2本鎖切断の修復過程では、切断部位周辺のクロマチンでリン酸化が起き、次に、シグナルタンパク質であるユビキチンが付加(ユビキチン化)された後に、それを目印として53BP1やBRCA1といった修復タンパク質が集積してくることがわかっています (図1) 。ユビキチンは、自身にユビキチンが付加することで数珠つなぎの鎖(ユビキチン鎖)を形成して機能する例が多く知られていますが、DNA2本鎖切断の修復過程でもユビキチン鎖が形成されます。DNA2本鎖切断の修復過程での損傷クロマチンのユビキチン化では、ユビキチン付加酵素であるRNF168が必須な役割を担い、53BP1やBRCA1に加えてRNF168自身もユビキチン鎖を目印として集積することがわかっていました。RNF168には、ユビキチンと結合する領域が複数知られていますが、それらのうちどの領域がどのようにユビキチン単独あるいはユビキチン鎖と結合するのか、また、どう使い分けがなされているのかは不明でした。
研究内容
これまでの研究により、損傷クロマチンの目印として63番目のリジン残基(K63)を介してつながったユビキチン鎖(K63鎖)が必要であることがわかっています。研究グループは、RNF168に含まれる既知のユビキチン結合ドメインに着目して、複数の生物種間でのアミノ酸配列の保存性も考慮に入れながら、精製タンパクの結合実験により、K63鎖選択的な結合に必要な領域を探りました。その結果、アミノ末端側に存在するUDM1と呼ぶ領域とカルボキシ末端側に存在するUDM2と呼ぶ二つの領域にK63鎖選択的な結合に必要な機能ユニットを同定しました。
UDM1には、UMIとMIU1という二つの直列したユビキチン結合ユニットが知られており、これまでは、この二つの結合ユニットが協働してK63鎖と選択的に結合すると考えられてきました。今回、研究グループの解析により、MIU1はK63鎖選択的な結合には不要であり、むしろ、UMIのN末端側に存在するLRMと呼ばれるユニットがUMIと協働してK63鎖と選択的に結合することが新たに判明しました。一方、UDM2はK63鎖に選択的に結合するにも関わらず、MIU2というユビキチン結合ユニットのみではK63鎖への選択性はないことがわかっていました。本研究において、研究グループは、UADと名付けた新たなユニットがMIU2と協働してK63鎖選択的に結合することを新たに見出しました。
さらに、大型放射光施設SPring-8(兵庫県佐用郡)の高輝度X線を利用してUDM1およびUDM2とK63鎖との複合体の結晶構造を解析し、UDM1中のLRM–UMIおよびUDM2中のUAD–MIU2がK63鎖と相互作用する様子を高解像度で明らかにしました。また、ヒト由来細胞株を用いた実験により、ユビキチン鎖との結合に必要なドメインの変異体はDNA損傷部位に蓄積しないことが示され、タンパク結合実験の結果や決定した立体構造が、生きた細胞でも当てはまることが実証されました。
社会的意義と今後の予定
RNF168は、K63鎖と相互作用するだけではなく、ヒストンタンパク質とも相互作用することが示唆されていますが、その詳細な仕組みはわかっていません。また、RNF168が触媒するヒストンのユビキチン化反応とK63鎖との結合の関連や、もう一つの重要なユビキチン付加酵素であるRNF8との機能的役割の違いなども立体構造に基づいた理解は明確にされていません。今後は、損傷クロマチン上でのRNF168の立体構造を知ることで、ヒストンのユビキチン化の反応状態を明らかにする研究が必要です。
特記事項
雑誌名:「Nature Communications」(オンライン版1月12日掲載)
論文タイトル:Structural insights into two distinct binding modules for Lys63-linked polyubiquitinchains in RNF168
著者:Tomio S. Takahashi,Yoshihiro Hirade, Aya Toma, Yusuke Sato, Atsushi Yamagata, SakurakoGoto-Ito, Akiko Tomita, Shinichiro Nakada* and Shuya Fukai*
DOI 番号:10.1038/s41467-017-02345-y
参考図
図1 DNA2本鎖切断の修復におけるRNF168の働き
参考URL
大阪大学 大学院医学系研究科 細胞応答制御学
http://www.bcr.med.osaka-u.ac.jp/public_html/index.html
用語説明
- RNF168
DNA2本鎖切断の修復に必須なタンパク質の一つ。DNA損傷部位の目印となるユビキチン(酵母からヒトまで保存された小さな球状タンパク質。シグナル分子として多様な細胞機能を制御する)を付加する酵素活性を持つ。
- ユビキチン鎖
ユビキチンは、基本的に標的タンパク質の特定のリジン残基に共有結合を介して付加される。ユビキチン自身も標的タンパク質の一つであり、ユビキチン同士が結合して数珠つなぎになったものをユビキチン鎖と呼ぶ。
